AIエンジニアが「R」を学ぶ必要があるのか?
もしあなたが、「Pythonですべて完結できるのに、なぜ今さらR言語なんて…」と思っているなら、少しだけ耳を傾けてください。
確かに、ディープラーニングや機械学習モデルの実装において、Python(PyTorchやTensorFlow)は無敵の王者です。しかし、医療AIの研究開発、特に「臨床研究」としての側面が強くなるフェーズでは、Pythonだけでは突破できない「壁」に直面することがあります。
その壁とは、「統計解析の厳密性」と「査読の壁」です。
医学統計の歴史において、R言語(およびそのパッケージ群)は長年にわたりデファクトスタンダードの地位を築いてきました。多くの医学論文で引用される統計手法や検定アルゴリズムは、Rのパッケージ(例えば survival や lme4 など)を基準に実装・検証されています。Pythonの scikit-survival や lifelines も素晴らしいライブラリですが、歴史あるRのパッケージが持つ機能の網羅性や、査読者からの信頼感には一日の長があります。
また、可視化においても、Rの ggplot2 が出力するグラフは、「論文にそのまま載せられる(Publication-Ready)」美しさとカスタマイズ性を誇り、多くの研究者を魅了してやみません。
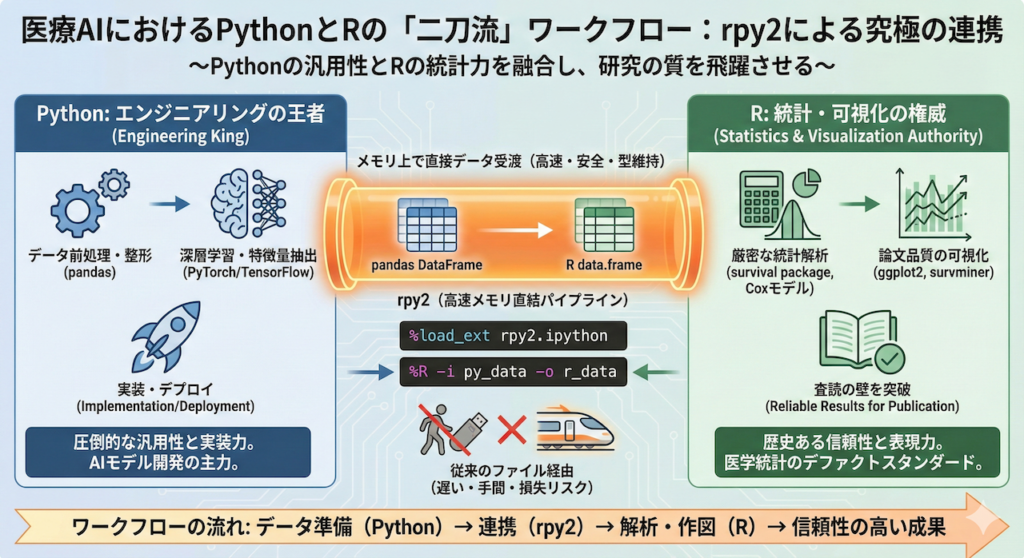
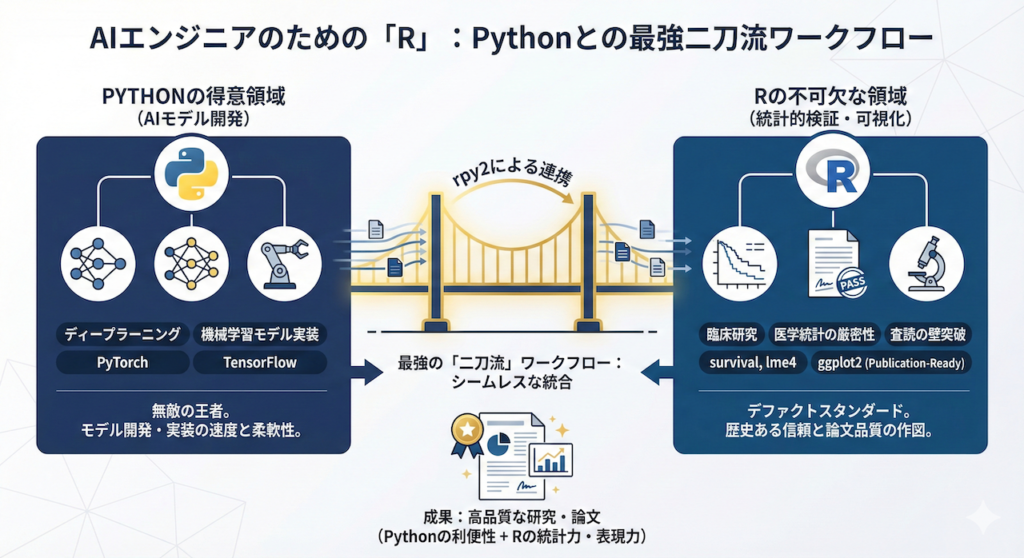
今回は、Pythonの利便性を手放すことなく、Rの強力な統計機能と表現力を手に入れるための架け橋、rpy2 を用いた連携術を伝授します。これは、AIモデル開発はPythonで、統計的検証と作図はRで行うという、最強の「二刀流」ワークフローへの招待状です。
1. 架け橋となる技術:「rpy2」とは何か?
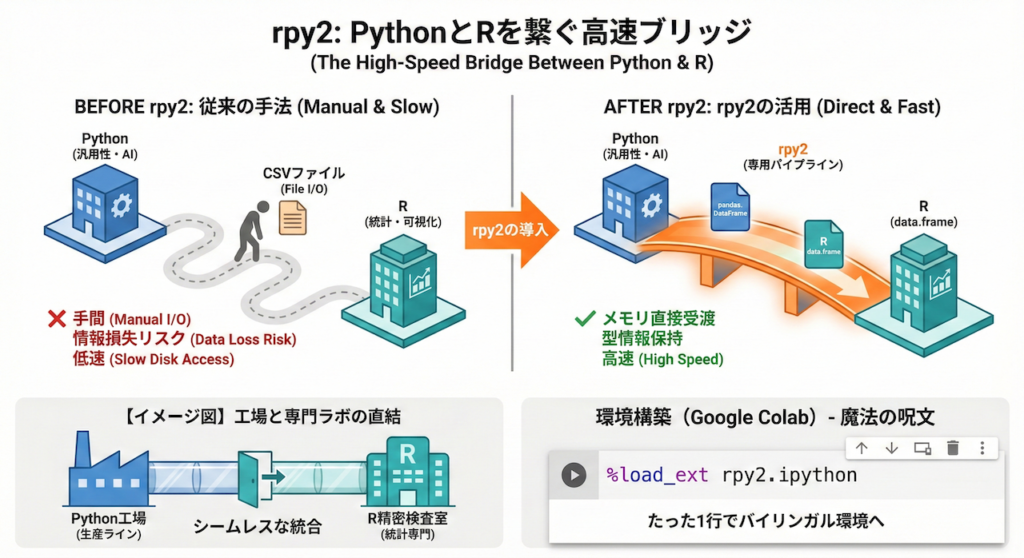
PythonとR、二つの巨人を繋ぐ「専用パイプライン」
データサイエンスの世界では長らく、「Python(汎用性・深層学習に強い)」と「R(統計解析・可視化に強い)」のどちらを使うべきかという議論がありました。しかし、医療AI開発の現場における正解は「両方使う」です。
rpy2 は、まさにそのためのツールです。これは、Pythonのプログラムの中から、R言語を直接呼び出して操作できるようにする強力なライブラリです。
通常、Pythonで整形したデータをRで解析しようとすると、一度CSVファイルなどに保存して書き出し、それをRで読み込み直すという手順が必要でした。これには以下のデメリットがあります:
- 手間がかかる: ファイルの入出力(I/O)処理をコードに書く必要がある。
- 情報が失われる: データの「型」情報(カテゴリー変数など)がCSV経由だと正しく引き継がれない場合がある。
- 遅い: ディスクへの書き込み・読み込みが発生するため処理速度が落ちる。
rpy2 を使えば、Pythonの pandas.DataFrame(データフレーム)を、メモリ上でそのまま Rの data.frame として受け渡すことができます。ファイルを経由せず、メモリ同士で直接データをやり取りするため、高速かつ安全です。
【イメージ図】
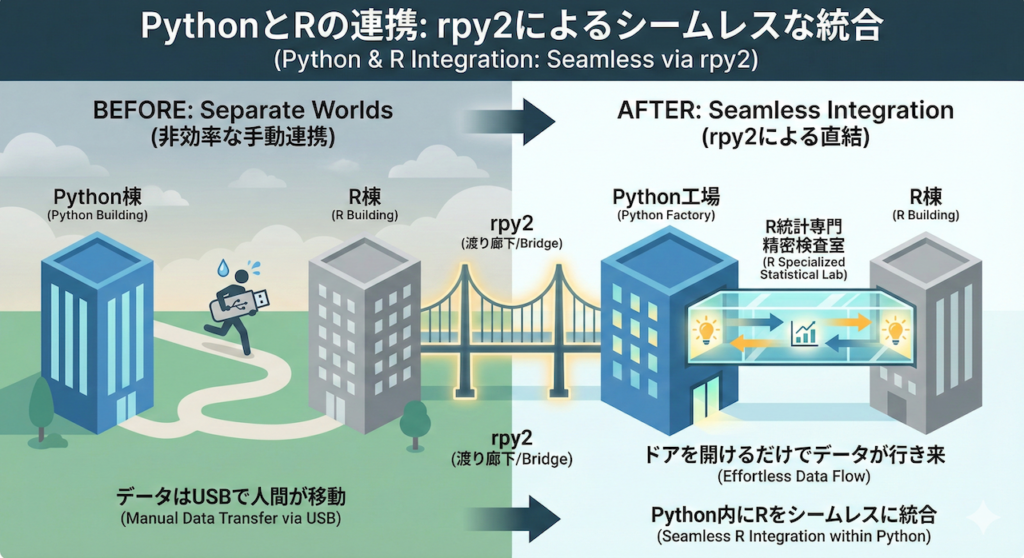
たとえるなら、これまでは「Python棟」と「R棟」が別々の建物で、データをUSBメモリに入れて人間が走って移動していた状態でした。rpy2 は、この二つの建物を渡り廊下で直結し、ドアを開けるだけでデータを行き来できるようにした状態と言えます。これにより、Pythonという工場の生産ラインの中に、Rという「統計専門の精密検査室」をシームレスに組み込むことが可能になるのです。
環境構築(Google Colab / Local)
それでは、実際にこの環境を作ってみましょう。
多くの医療従事者や初学者が利用する Google Colab には、実はすでに rpy2 がプリインストールされています。そのため、複雑なインストール作業は不要で、魔法の呪文(マジックコマンド)を唱えるだけで準備が完了します。
以下のコードを、Google Colabの最初のセルに入力して実行してください。
# Google Colabなどで実行する場合
# Pythonのノートブック内でRを使えるようにする拡張機能を読み込む
%load_ext rpy2.ipython解説:
%load_ext: Jupyter Notebook(Colab)の拡張機能をロードするコマンドです。rpy2.ipython: Pythonの対話環境(IPython)でRを使えるようにするモジュールです。
たったこの一行を実行するだけで、あなたのノートブックはPythonとRのバイリンガル環境へと進化します。これ以降、セルの冒頭に %%R と書くだけで、そのセル内ではR言語のコードを自由に記述・実行できるようになります。
2. 実践:生存時間分析における「二刀流」ワークフロー
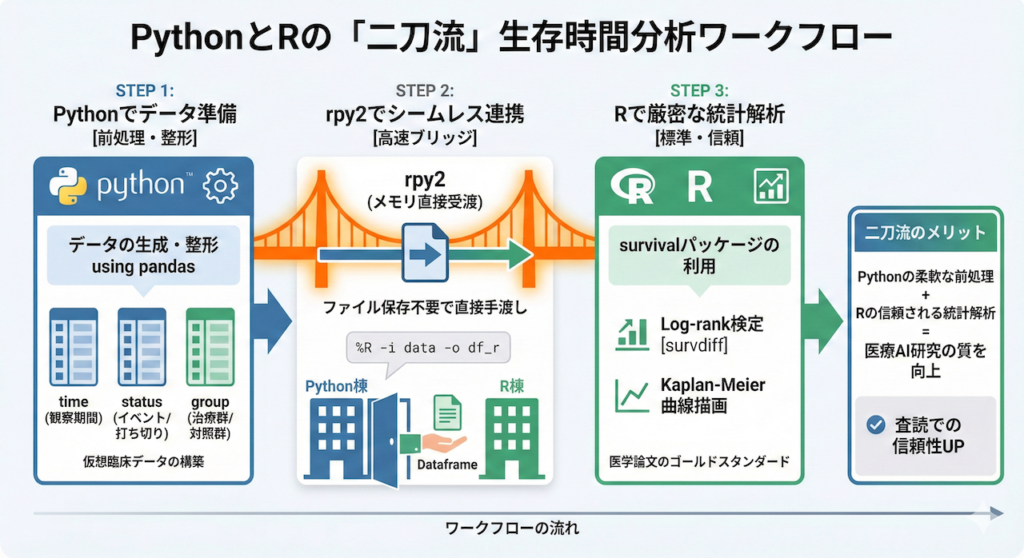
では、具体的なシナリオで「PythonとRのいいとこ取り」を体験してみましょう。
医療AI開発において、患者さんの予後(生存期間や再発までの期間)を予測するタスクは極めて重要です。ここでは、以下の役割分担で、シームレスな解析フローを構築します。
- Python (前処理): データの生成、整形、クレンジング(pandasなどを使用)
- R (統計解析): 統計的に厳密なKaplan-Meier曲線の描画と、Log-rank検定(survivalパッケージを使用)
ステップ1:Pythonでデータを準備する
まずは、Python側で仮想的な臨床データを生成し、pandas データフレームとして管理します。ここでは、新しい治療法(Treatment)と従来の治療法(Control)を比較する、肺がん患者さんの予後データをシミュレーションしてみます。
解説:
以下のコードでは、「観察期間(time)」と「イベント発生状況(status)」、そして「群(group)」を作成しています。
status: 1 はイベント発生(死亡など)、0 は打ち切り(転院や生存確認など)を表します。生存時間分析特有の「打ち切り(Censoring)」データを含めるのがポイントです。
import pandas as pd
import numpy as np
# 再現性のために乱数シードを固定
np.random.seed(42)
# サンプル数
n = 100
# 仮想データの生成(肺がん患者の予後データを想定)
data = pd.DataFrame({
# 指数分布と正規分布を組み合わせて生存時間をシミュレーション
'time': np.random.exponential(10, n) + np.random.normal(0, 1, n).clip(min=0),
# 0:打ち切り, 1:イベント発生(死亡)
# 確率的に打ち切りを含める(ここでは70%がイベント発生と仮定)
'status': np.random.choice([0, 1], n, p=[0.3, 0.7]),
# 治療群と対照群をランダムに割り当て
'group': np.random.choice(['Treatment', 'Control'], n)
})
# 治療群(Treatment)の方が、対照群より少し長生きするようにデータを調整
data.loc[data['group'] == 'Treatment', 'time'] += 5
# データの確認
print(data.head()) time status group
0 4.779728 1 Control
1 35.101214 1 Treatment
2 18.259218 1 Treatment
3 9.129426 1 Control
4 6.696249 0 Treatmentステップ2:PythonからRへデータを渡す
ここで rpy2 の真骨頂です。Pythonのデータを、ファイル保存することなく、メモリ上で直接Rに手渡します。
マジックコマンド %R を使用し、オプション引数でデータの受け渡しを指示します。
-i(input): PythonからRへ渡す変数(ここではdata)-o(output): RからPythonへ戻す変数(ここではdf_r)
# pandasのDataFrame 'data' を Rの変数 'df_r' として転送し、
# その結果をPython側の変数 'df_r' としても受け取る
%R -i data -o df_r df_r <- data time status group
0 4.779728 1 Control
1 35.101214 1 Treatment
2 18.259218 1 Treatment
3 9.129426 1 Control
4 6.696249 0 Treatment
... ... ... ...
95 6.808147 1 Control
96 7.396788 1 Control
97 10.919294 1 Treatment
98 5.534169 1 Treatment
99 6.968858 0 Treatment
100 rows × 3 columnsたったこれだけの記述で、Pythonの pandas.DataFrame が、Rの data.frame に自動変換されて渡されました。まるで「Python棟」と「R棟」の間のドアを開けて、書類を手渡ししたようなスムーズさです。
ステップ3:Rによる厳密な統計解析(Log-rank検定)
データがR側に渡ったので、ここからはRの独壇場です。
セル全体をRのコードとして実行する %%R マジックコマンドを使い、生存時間分析のゴールドスタンダードである survival パッケージ を呼び出します。
ここで行うのは以下の処理です:
Surv(): 生存時間とイベント情報を組み合わせた「生存オブジェクト」を作成する。survdiff(): 群間(治療群 vs 対照群)で生存率に差があるかを検定する(Log-rank検定)。
%%R
# Rのライブラリを読み込み(survivalはRに標準で含まれることが多い強力なパッケージです)
library(survival)
# 生存オブジェクトの作成
# time: 観察期間, event: イベント発生状況(1=発生, 0=打ち切り)
surv_obj <- Surv(time = df_r$time, event = df_r$status)
# Log-rank検定の実行
# group(群)によって生存曲線に差があるかを検定
fit_diff <- survdiff(surv_obj ~ group, data = df_r)
# 検定結果の表示
print(fit_diff)
# カイ二乗値からp値を計算して表示
p_value <- 1 - pchisq(fit_diff$chisq, length(fit_diff$n) - 1)
cat("\nLog-rank test p-value:", p_value, "\n")Call:
survdiff(formula = surv_obj ~ group, data = df_r)
N Observed Expected (O-E)^2/E (O-E)^2/V
group=Control 55 40 35.8 0.492 0.972
group=Treatment 45 33 37.2 0.474 0.972
Chisq= 1 on 1 degrees of freedom, p= 0.3
Log-rank test p-value: 0.3242257 なぜこれが重要なのか?
Pythonのライブラリ(lifelines など)も優秀ですが、医学論文の査読プロセスにおいては、歴史と実績のある Rの survival パッケージ で検証された結果の方が、査読者にとって馴染み深く、信頼されやすい傾向があります。「Pythonでモデルを作り、Rで統計的裏付けを取る」。この二刀流こそが、医療AI研究の質を高める賢い戦略なのです。
3. ggplot2による「出版品質」の可視化
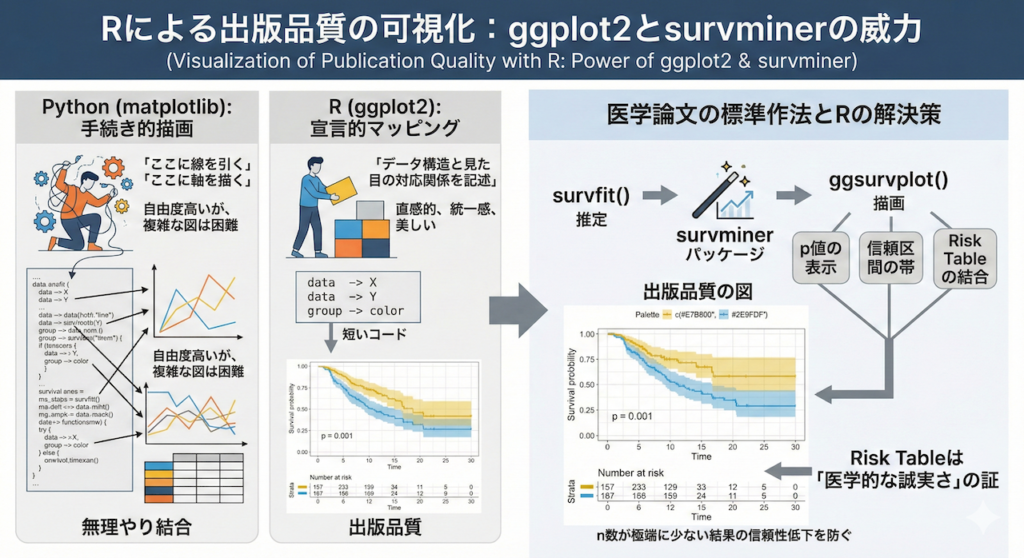
なぜ、論文の図は「R」なのか?
R言語を使うもう一つの、そして最大の理由が ggplot2 です。
Pythonにも matplotlib や seaborn といった優れた可視化ライブラリがありますが、Rの ggplot2 が持つ「Grammar of Graphics(グラフィックスの文法)」という設計思想は、学術界において依然として圧倒的な支持を得ています。
- Python (matplotlib): 「ここに線を引く」「ここに軸を描く」というように、手続き的に描画を指示します。自由度は高いですが、複雑な図を作るにはコードが長くなりがちです。
- R (ggplot2): 「データAをX軸に、データBをY軸に、グループごとに色を変えて」というように、データの構造と見た目の対応関係(マッピング)を記述します。これにより、直感的かつ統一感のある美しい図を、短いコードで作成できます。
魔法のパッケージ「survminer」
特に、医学論文(生存時間分析)においては、グラフの下に「Risk Table(No. at risk: 観察期間ごとの生存者数テーブル)」を付記することが、事実上の標準ルール(作法)となっています。
これをPythonだけで美しく実装しようとすると、複数のグラフを無理やり結合するような複雑なコーディングが必要になりますが、Rのエコシステムでは、まさにそのための専用ツール survminer が用意されています。
準備:パッケージのインストール
survminer は標準パッケージではないため、使用する環境(Google Colabなど)によっては追加でインストールする必要があります。以下のコマンドを実行して、環境にパッケージを追加しましょう。
%%R
# パッケージのインストール(初回のみ必要)
# ミラーサイトを指定してインストールします
install.packages("survminer", repos = "https://cloud.r-project.org")実践:Publication-Readyな図を描く
準備ができたら、実際に描画してみましょう。以下のコードでは、「p値の表示」「信頼区間の帯」「Risk Tableの結合」といった、論文に必要な要素をすべて含んだグラフを、たった数行のコマンドで作成します。
【コード解説】
survfit(): 生存曲線の推定を行います(ここではKaplan-Meier法)。ggsurvplot(): 推定結果をもとに、グラフを描画します。引数を指定するだけで、プロ並みの図が出来上がります。
%%R
# 必要なライブラリの読み込み
library(survminer)
library(ggplot2)
library(survival)
# 1. Kaplan-Meier推定の実行
# time: 観察期間, status: イベント発生状況
# ~ group: 群(治療群 vs 対照群)ごとに層別化する
fit <- survfit(Surv(time, status) ~ group, data = df_r)
# 2. 美しいグラフの描画
ggsurv <- ggsurvplot(
fit, # 推定したモデル
data = df_r, # 使用するデータ
pval = TRUE, # グラフ内にp値(検定結果)を表示する
conf.int = TRUE, # 95%信頼区間(帯)を表示する
risk.table = TRUE, # グラフの下にRisk Table(生存数)を表示する
risk.table.col = "strata", # Risk Tableの文字色を群ごとに変える
palette = c("#E7B800", "#2E9FDF"), # グラフの色(黄色と青)を指定
ggtheme = theme_bw(), # 背景を白にする(論文掲載用のクリーンなテーマ)
main = "Kaplan-Meier Curve via R (rpy2)" # グラフのタイトル
)
# 描画
print(ggsurv)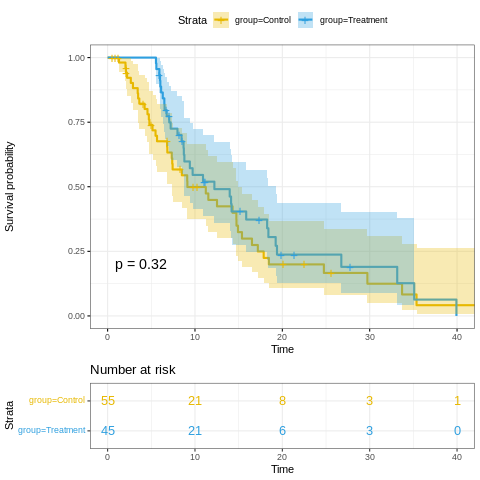
【出力結果のイメージ】
このコードを実行すると、上段に生存曲線(生存率の推移)、下段にRisk Table(その時点で観察対象となっている患者数)が綺麗に整列した図が出力されます。
特に risk.table = TRUE は重要です。グラフの右端(長期生存)で生存率が高く見えても、もしRisk Tableの人数(n数)が極端に少なければ、その結果は信頼性が低い可能性があるからです。この情報をセットで提示することが、医学的な誠実さ(Scientific Integrity)の証となります。
4. 厳密な統計解析への接続:Cox比例ハザードモデル
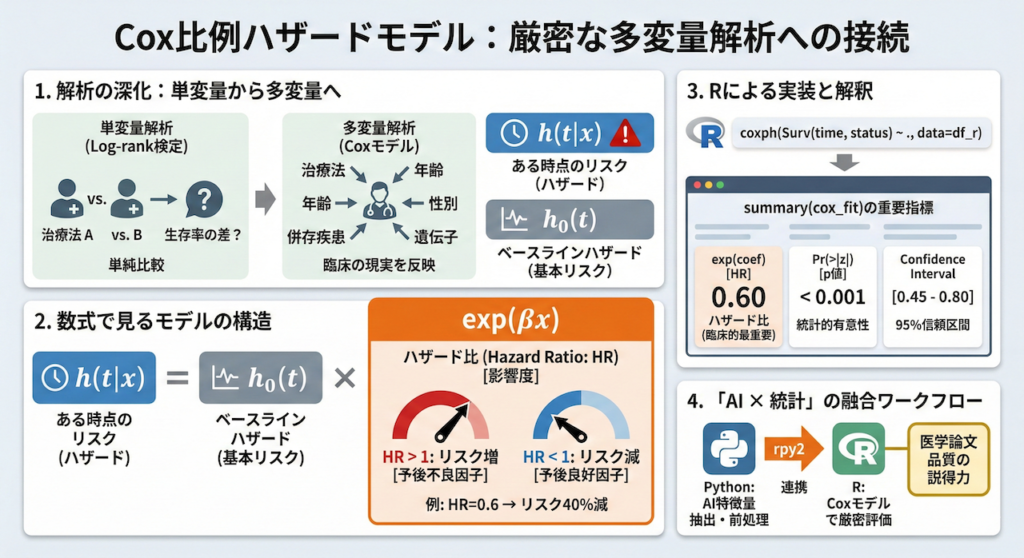
単変量から多変量へ:臨床の現実に即した解析
先ほどのLog-rank検定は、2つの群(治療群 vs 対照群)の間に生存率の差があるかどうかを見る「単変量解析」でした。しかし、実際の臨床現場では、患者さんの予後を左右するのは治療法の違いだけではありません。年齢、性別、併存疾患、遺伝子変異など、様々な要因(共変量)が複雑に絡み合っています。
これらの要因をすべて考慮した上で、「純粋にこの治療法に効果があるのか?」、あるいは「どの因子がリスクを高めているのか?」を解き明かすために使われるのが、Cox比例ハザードモデル(多変量解析)です。
Pythonの lifelines パッケージでも実行可能ですが、Rの coxph 関数は、変数の選択、モデルの診断(比例ハザード性の検証など)、結果の要約出力において、非常に洗練されており、医学論文の標準フォーマットに即した結果を即座に得ることができます。
数式で見るモデルの構造
統計学的な背景を少しだけ掘り下げましょう。Cox比例ハザードモデルでは、ある時点 \( t \) におけるイベント発生のリスク(ハザード関数 \( h(t) \))を、以下のようにモデル化します。
\[ h(t|x) = h_0(t) \exp(\beta_1 x_1 + \beta_2 x_2 + \dots + \beta_p x_p) \]
ここで、各項は以下の意味を持ちます。
- \( h(t|x) \): 共変量 \( x \) を持つ個体の、時点 \( t \) におけるハザード(瞬間死亡率)。
- \( h_0(t) \): ベースラインハザード関数。すべての共変量が0のときのハザードを表します。Coxモデルの凄いところは、この関数の形状を特定しなくても解析ができる点(セミパラメトリック手法)にあります。
- \( \exp(\beta_i) \): これがハザード比(Hazard Ratio: HR)に対応します。
- \( \exp(\beta_i) > 1 \): その因子が増えるとリスクが上がる(予後不良因子)。
- \( \exp(\beta_i) < 1 \): その因子が増えるとリスクが下がる(予後良好因子)。
Rによる実装と解釈
それでは、rpy2 を介してRの coxph 関数を実行してみましょう。Python側で前処理した特徴量をRに渡し、Rの強力なエンジンで計算させます。
%%R
# Cox比例ハザードモデルのあてはめ
# time: 観察期間, status: イベント
# group: 解析したい因子(ここでは治療群か対照群か)
cox_fit <- coxph(Surv(time, status) ~ group, data = df_r)
# 結果の要約を表示
summary(cox_fit)Call:
coxph(formula = Surv(time, status) ~ group, data = df_r)
n= 100, number of events= 73
coef exp(coef) se(coef) z Pr(>|z|)
groupTreatment -0.2320 0.7929 0.2359 -0.984 0.325
exp(coef) exp(-coef) lower .95 upper .95
groupTreatment 0.7929 1.261 0.4994 1.259
Concordance= 0.56 (se = 0.033 )
Likelihood ratio test= 0.97 on 1 df, p=0.3
Wald test = 0.97 on 1 df, p=0.3
Score (logrank) test = 0.97 on 1 df, p=0.3
この summary(cox_fit) を実行すると、以下のような重要な指標が一括で出力されます。
- coef: 回帰係数 \( \beta \)。
- exp(coef): ハザード比(HR)。臨床的に最も重要な値です。例えば、治療群のHRが0.6であれば、「治療群は対照群に比べて死亡リスクが40%低い」と解釈できます。
- Pr(>|z|): p値。その因子が統計的に有意かどうかを判断します。
- Confidence Interval: ハザード比の95%信頼区間。
「AI × 統計」の融合ワークフロー
ここまでの流れを振り返ると、私たちのワークフローは以下のようになります。
- Python: 複雑なデータクレンジング、深層学習による特徴量抽出(画像からリスクスコアを算出するなど)。
- R: 抽出された特徴量をCoxモデルに投入し、医学的に解釈可能な形(ハザード比)で評価する。
これこそが、最先端のAI技術と、伝統的かつ厳密な統計学を融合させる「データサイエンティスト」の働き方です。PythonとR、それぞれの得意分野を活かすことで、研究の説得力は格段に増すことでしょう。
まとめ:道具を選ばない、真の「データサイエンティスト」へ
「Python vs R」ではなく「Python and R」
長きにわたり、データサイエンス界隈では「PythonとR、どちらを学ぶべきか?」という議論が繰り返されてきました。しかし、医療AIという高度で複合的な領域においては、その議論はもはや過去のものです。
真に実力のあるデータサイエンティストや、臨床現場に変革をもたらす医療AI研究者は、「PythonもRも」使いこなします。それは、大工が金槌と鋸(のこぎり)を使い分けるように、目的に応じて最適な道具を手に取る「二刀流」のスタイルです。
最適な役割分担:適材適所の哲学
本記事で実践したように、それぞれの言語には明確な強みがあります。
- Python (エンジニアリングの王者):
- 役割: データの前処理、深層学習モデル(PyTorch/TensorFlow)の構築、API化、システムへのデプロイ。
- 強み: 圧倒的な汎用性と、本番環境への実装力。
- R (統計と可視化の権威):
- 役割: 探索的データ解析(EDA)、厳密な統計検定(生存時間分析など)、論文投稿用の高品質な作図(ggplot2)。
- 強み: アカデミアで培われた統計的な信頼性と、表現力の豊かさ。
この役割分担を深く理解し、rpy2 という架け橋を自由に渡れるようになること。それは単に「使えるツールが増える」以上の意味を持ちます。あなたの研究結果に対する質(Quality)と、医学的な信頼性(Reliability)が飛躍的に向上することを意味するのです。
参考文献
- Cox, D.R. (1972). Regression Models and Life-Tables. Journal of the Royal Statistical Society: Series B (Methodological), 34(2), 187–202.
- Gautier, L. (2024). rpy2: Python interface to the R language. Available at: https://rpy2.github.io/
- Kaplan, E.L. and Meier, P. (1958). Nonparametric Estimation from Incomplete Observations. Journal of the American Statistical Association, 53(282), 457–481.
- Kassambara, A., Kosinski, M. and Biecek, P. (2021). survminer: Drawing Survival Curves using ‘ggplot2’. R package version 0.4.9. Available at: https://CRAN.R-project.org/package=survminer
- Mantel, N. (1966). Evaluation of Survival Data and Two New Rank Order Statistics Arising in Its Consideration. Cancer Chemotherapy Reports, 50(3), 163–170.
- R Core Team (2024). R: A Language and Environment for Statistical Computing. R Foundation for Statistical Computing, Vienna, Austria. Available at: https://www.R-project.org/
- Therneau, T.M. (2024). A Package for Survival Analysis in R. R package version 3.5-8. Available at: https://CRAN.R-project.org/package=survival
- Wickham, H. (2016). ggplot2: Elegant Graphics for Data Analysis. Springer-Verlag New York.
※本記事は情報提供を目的としたものであり、特定の治療法を推奨するものではありません。健康に関するご懸念やご相談は、必ず専門の医療機関にご相談ください。
ご利用規約(免責事項)
当サイト(以下「本サイト」といいます)をご利用になる前に、本ご利用規約(以下「本規約」といいます)をよくお読みください。本サイトを利用された時点で、利用者は本規約の全ての条項に同意したものとみなします。
第1条(目的と情報の性質)
- 本サイトは、医療分野におけるAI技術に関する一般的な情報提供および技術的な学習機会の提供を唯一の目的とします。
- 本サイトで提供されるすべてのコンテンツ(文章、図表、コード、データセットの紹介等を含みますが、これらに限定されません)は、一般的な学習参考用であり、いかなる場合も医学的な助言、診断、治療、またはこれらに準ずる行為(以下「医行為等」といいます)を提供するものではありません。
- 本サイトのコンテンツは、特定の製品、技術、または治療法の有効性、安全性を保証、推奨、または広告・販売促進するものではありません。紹介する技術には研究開発段階のものが含まれており、その臨床応用には、さらなる研究と国内外の規制当局による正式な承認が別途必要です。
- 本サイトは、情報提供を目的としたものであり、特定の治療法を推奨するものではありません。健康に関するご懸念やご相談は、必ず専門の医療機関にご相談ください。
第2条(法令等の遵守)
利用者は、本サイトの利用にあたり、医師法、医薬品、医療機器等の品質、有効性及び安全性の確保等に関する法律(薬機法)、個人情報の保護に関する法律、医療法、医療広告ガイドライン、その他関連する国内外の全ての法令、条例、規則、および各省庁・学会等が定める最新のガイドライン等を、自らの責任において遵守するものとします。これらの適用判断についても、利用者が自ら関係各所に確認するものとし、本サイトは一切の責任を負いません。
第3条(医療行為における責任)
- 本サイトで紹介するAI技術・手法は、あくまで研究段階の技術的解説であり、実際の臨床現場での診断・治療を代替、補助、または推奨するものでは一切ありません。
- 医行為等に関する最終的な判断、決定、およびそれに伴う一切の責任は、必ず法律上その資格を認められた医療専門家(医師、歯科医師等)が負うものとします。AIによる出力を、資格を有する専門家による独立した検証および判断を経ずに利用することを固く禁じます。
- 本サイトの情報に基づくいかなる行為によって利用者または第三者に損害が生じた場合も、本サイト運営者は一切の責任を負いません。実際の臨床判断に際しては、必ず担当の医療専門家にご相談ください。本サイトの利用によって、利用者と本サイト運営者の間に、医師と患者の関係、またはその他いかなる専門的な関係も成立するものではありません。
第4条(情報の正確性・完全性・有用性)
- 本サイトは、掲載する情報(数値、事例、ソースコード、ライブラリのバージョン等)の正確性、完全性、網羅性、有用性、特定目的への適合性、その他一切の事項について、何ら保証するものではありません。
- 掲載情報は執筆時点のものであり、予告なく変更または削除されることがあります。また、技術の進展、ライブラリの更新等により、情報は古くなる可能性があります。利用者は、必ず自身で公式ドキュメント等の最新情報を確認し、自らの責任で情報を利用するものとします。
第5条(AI生成コンテンツに関する注意事項)
本サイトのコンテンツには、AIによる提案を基に作成された部分が含まれる場合がありますが、公開にあたっては人間による監修・編集を経ています。利用者が生成AI等を用いる際は、ハルシネーション(事実に基づかない情報の生成)やバイアスのリスクが内在することを十分に理解し、その出力を鵜呑みにすることなく、必ず専門家による検証を行うものとします。
第6条(知的財産権)
- 本サイトを構成するすべてのコンテンツに関する著作権、商標権、その他一切の知的財産権は、本サイト運営者または正当な権利を有する第三者に帰属します。
- 本サイトのコンテンツを引用、転載、複製、改変、その他の二次利用を行う場合は、著作権法その他関連法規を遵守し、必ず出典を明記するとともに、権利者の許諾を得るなど、適切な手続きを自らの責任で行うものとします。
第7条(プライバシー・倫理)
本サイトで紹介または言及されるデータセット等を利用する場合、利用者は当該データセットに付随するライセンス条件および研究倫理指針を厳格に遵守し、個人情報の匿名化や同意取得の確認など、適用される法規制に基づき必要とされるすべての措置を、自らの責任において講じるものとします。
第8条(利用環境)
本サイトで紹介するソースコードやライブラリは、執筆時点で特定のバージョンおよび実行環境(OS、ハードウェア、依存パッケージ等)を前提としています。利用者の環境における動作を保証するものではなく、互換性の問題等に起因するいかなる不利益・損害についても、本サイト運営者は責任を負いません。
第9条(免責事項)
- 本サイト運営者は、利用者が本サイトを利用したこと、または利用できなかったことによって生じる一切の損害(直接損害、間接損害、付随的損害、特別損害、懲罰的損害、逸失利益、データの消失、プログラムの毀損等を含みますが、これらに限定されません)について、その原因の如何を問わず、一切の法的責任を負わないものとします。
- 本サイトの利用は、学習および研究目的に限定されるものとし、それ以外の目的での利用はご遠慮ください。
- 本サイトの利用に関連して、利用者と第三者との間で紛争が生じた場合、利用者は自らの費用と責任においてこれを解決するものとし、本サイト運営者に一切の迷惑または損害を与えないものとします。
- 本サイト運営者は、いつでも予告なく本サイトの運営を中断、中止、または内容を変更できるものとし、これによって利用者に生じたいかなる損害についても責任を負いません。
第10条(規約の変更)
本サイト運営者は、必要と判断した場合、利用者の承諾を得ることなく、いつでも本規約を変更することができます。変更後の規約は、本サイト上に掲載された時点で効力を生じるものとし、利用者は変更後の規約に拘束されるものとします。
第11条(準拠法および合意管轄)
本規約の解釈にあたっては、日本法を準拠法とします。本サイトの利用および本規約に関連して生じる一切の紛争については、東京地方裁判所を第一審の専属的合意管轄裁判所とします。
For J³, may joy follow you.