基本構造から、データ不足を克服する実践テクニックまで。
①畳み込み:特徴(エッジ・形)を抽出。
②ReLU:不要なノイズを遮断。
③Pooling:情報を要約し頑健性を確保。
- LeNet:CNNの始祖。
- AlexNet:深層学習ブームの点火。
- VGG:3×3フィルタの単純さと美学。
- ResNet:100層超えを可能にした革命。
データ拡張で水増しし、転移学習(既学習モデルの流用)で巨人の肩に乗る。
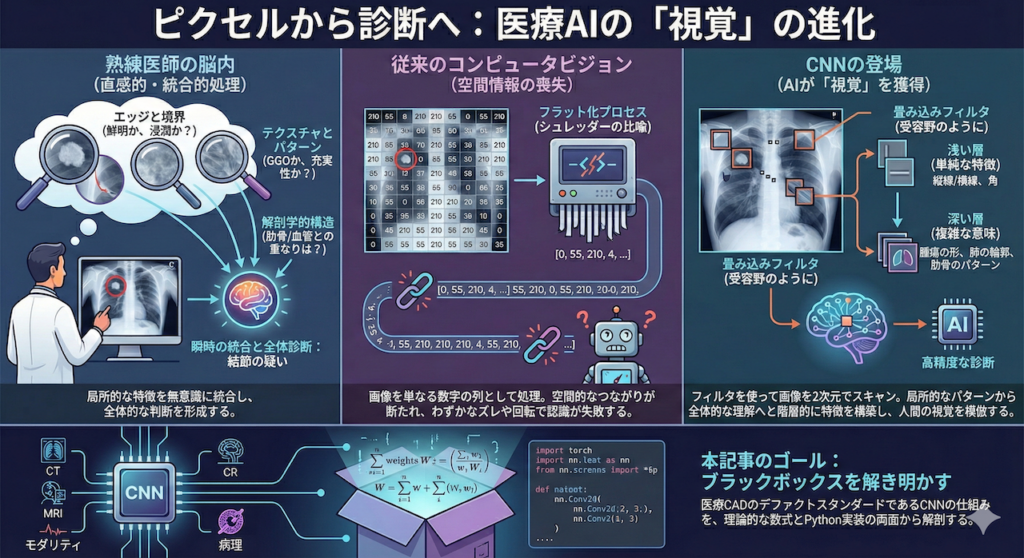
熟練した医師が胸部X線写真(CXR)を見て、「右肺上葉に結節の疑いがある」と瞬時に判断するとき、その脳内では一体何が起きているのでしょうか?
おそらく、医師は画像に含まれる数百万個の画素(ピクセル)一つひとつの「輝度値(明るさ)」を単純に計算しているわけではありません。そうではなく、以下のような高度な視覚的特徴処理を無意識のうちに行っているはずです。
- エッジと境界: 白い影の輪郭は鮮明か、あるいは周囲に浸潤しているか。
- テクスチャとパターン: すりガラス状の陰影(GGO)か、充実性の陰影か。
- 解剖学的構造: 血管や肋骨の走行と重なっていないか、肺野全体の構造に歪みはないか。
人間は、こうした「局所的な特徴」を瞬時に捉え、それらを脳内で統合することで「全体的な診断」を導き出しています。しかし、この「当たり前」のプロセスをコンピュータに再現させることは、長らく極めて困難な課題でした。
従来のコンピュータが見ていた「世界」
従来のコンピュータや古典的なニューラルネットワークにとって、画像は単なる「数字の羅列(グリッド)」に過ぎませんでした。例えば、28×28ピクセルの画像は、単に784個の数字が並んだ1列のデータとして処理されていました。
このアプローチでは、画像内のオブジェクトが少し右にずれたり、回転したりしただけで、データとしては「全く別の数字の列」になってしまい、認識精度が著しく低下するという欠点がありました。
CNNの登場:AIが「視覚」を獲得した日
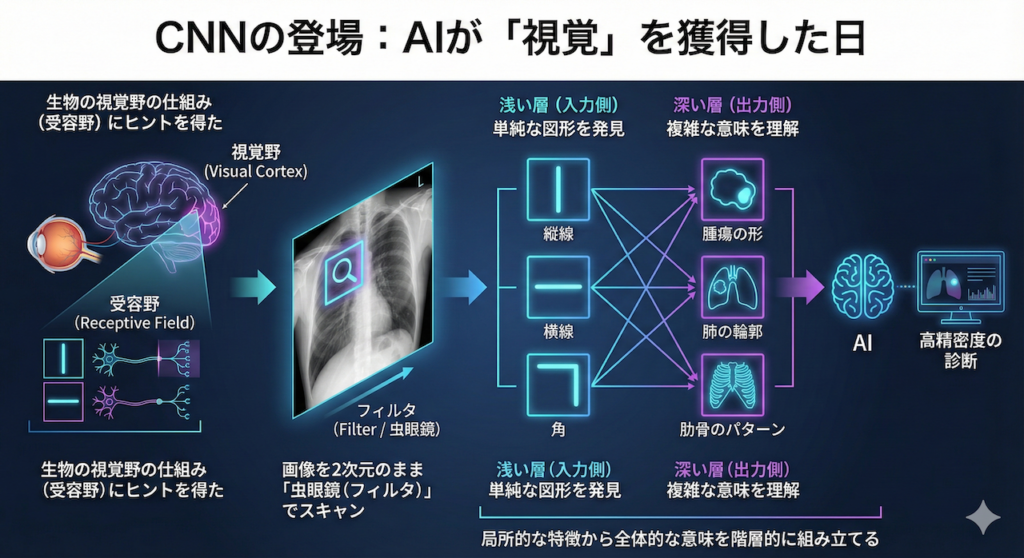
この壁を打ち破ったのが、畳み込みニューラルネットワーク(CNN: Convolutional Neural Network)です。CNNは、生物の視覚野にある「受容野(Receptive Field)」の仕組み——特定の角度の線や動きにだけ反応する細胞があること——にヒントを得て開発されました (Hubel & Wiesel 1962; LeCun et al. 1998)。
CNNは、画像を1列の数字にするのではなく、画像のまま(2次元のまま)小さな「虫眼鏡(フィルタ)」を通してスキャンします。
- 浅い層(入力に近い側): 「縦線」「横線」「角」といった単純な図形を発見する。
- 深い層(出力に近い側): それらを組み合わせて「目」「腫瘍の形」「肺の輪郭」といった複雑な意味を理解する。
このように、「局所的な特徴」から「全体的な意味」を階層的に組み立てるプロセスこそが、AIに人間並み、あるいはそれ以上の画像認識能力を与えたのです (Litjens et al. 2017)。
本記事のゴール
現在の医療AI診断支援システム(CAD: Computer-Aided Diagnosis)のほとんどは、このCNNを根幹技術としています。CT、MRI、病理画像、眼底写真など、モダリティを問わず、CNNは医療画像解析のデファクトスタンダード(事実上の標準)です。
今回は、この「AIの目」であるCNNの仕組みを、ブラックボックスにせず、数式による理論的背景とPythonによる実装の両面から徹底的に紐解きます。実際に手を動かしながら、単純な数字の羅列から「診断」が生まれる瞬間を体感してみましょう。
1. 畳み込みニューラルネットワーク(CNN)の解剖学
従来のニューラルネットワーク(全結合層)で画像を扱おうとすると、例えば「猫」が画像の左隅にいても右隅にいても同じ「猫」であると認識させるために、膨大なデータを学習させる必要がありました。これは、画像を1列の数字に変換してしまうことで、ピクセル同士の「上下左右のつながり(空間情報)」が失われてしまうからです。
この課題を解決したのが **CNN(Convolutional Neural Network)** です。CNNは、生物の視覚野にある「受容野(Receptive Field)」の構造——視野の一部だけを重点的に見る細胞が集まって全体像を構成する仕組み——を模倣して設計されました (LeCun et al. 1998)。
CNNは、主に以下の3つの「臓器(レイヤー)」が連携して機能します。
① 畳み込み層(Convolutional Layer):特徴を抽出する「フィルタ」
これがCNNの心臓部です。ここでは、画像全体を一度に見るのではなく、**「フィルタ(カーネル)」**と呼ばれる小さな窓をスライドさせながら、局所的な特徴をスキャンしていきます。
【直感的なイメージ:医師の読影】
レントゲン写真を見る医師を想像してください。医師は写真全体をぼんやり見るのではなく、「肺の右上に異常な白い影はないか?」「肋骨のラインは正常か?」と、視点を動かしながら特定の特徴(パターン)を探します。畳み込み層における「フィルタ」とは、まさにこの「特定の特徴(例:丸い結節、線状の骨折線)を探すための型枠」のようなものです。
【数式と計算の仕組み】
数学的には、入力画像 \(I\) とフィルタ \(K\) の「積和演算(対応する位置の数字を掛けて、全部足す)」を行います。これを畳み込み演算 \(S\) と呼びます。
\[ S(i, j) = (I * K)(i, j) = \sum_{m} \sum_{n} I(i – m, j – n) K(m, n) \]
数式だと難しく見えますが、やっていることは単純な掛け算と足し算です。以下の具体例を見てみましょう。
※ この値が大きいほど、「その場所に探している形(×印)があった」ことを意味します。
AIの学習が進むにつれて、この「フィルタの値」が自動的に調整され、最初は「縦線・横線」などの単純な形を、層が深くなるにつれて「腫瘍の形」や「臓器の輪郭」といった高度な特徴を検出できるように進化していきます。
② 活性化関数(Activation Function):非線形性の獲得
畳み込み演算(掛け算と足し算)だけでは、どれだけ層を重ねても「線形変換(直線のグラフ)」の組み合わせにしかならず、複雑な病変の形状や境界線を表現できません。そこで、「ここぞ」という信号だけを通すためのゲート役が必要になります。
現在、デファクトスタンダード(事実上の標準)として用いられるのが **ReLU (Rectified Linear Unit)** です (Nair & Hinton 2010)。
\[ f(x) = \max(0, x) \]
【直感的なイメージ:ニューロンの発火】
これは、脳の神経細胞(ニューロン)の挙動に似ています。
「入力が一定以下(0以下)なら反応しない(0を出力)。一定以上なら、その強さをそのまま伝える」
という非常にシンプルな仕組みです。
- 負の値(重要でない信号): 0にして切り捨てる(ノイズの除去)。
- 正の値(重要な特徴): そのまま次の層へ伝える。
この単純な操作により、ネットワークは「反応する/しない」という非線形な表現力を手に入れ、勾配消失問題(学習が途中で止まってしまう現象)も防ぐことができます。
③ プーリング層(Pooling Layer):情報を要約する
畳み込み層で抽出された特徴マップは、ピクセル単位の細かすぎる情報を含んでいます。「腫瘍がある」ことが重要なのであって、「腫瘍が右に1ピクセルずれている」ことは診断において重要ではありません。そこで、情報を圧縮・要約する処理を行います。
代表的なのが Max Pooling です。
【この操作の意義】
- 情報の要約: データ量が4分の1(上記例の場合)になり、計算コストが下がります。
- 頑健性(Robustness)の向上: 2×2の領域内のどこかに特徴(高い値)があれば、それが抽出されます。これにより、画像内で対象物が多少ズレたり回転したりしても、AIは「同じ特徴がある」と認識できるようになります(位置不変性)。
2. CNNアーキテクチャの進化:LeNetからVGG、そして深層の果てへ
CNNの歴史を紐解くことは、人類がコンピュータに「視覚」という知能を与えようとした、半世紀にわたる挑戦の歴史を追体験することに他なりません。それは、計算資源の制約と闘いながら、「より深く(Deeper)、より賢く」あろうとしたアルゴリズムの進化論です。
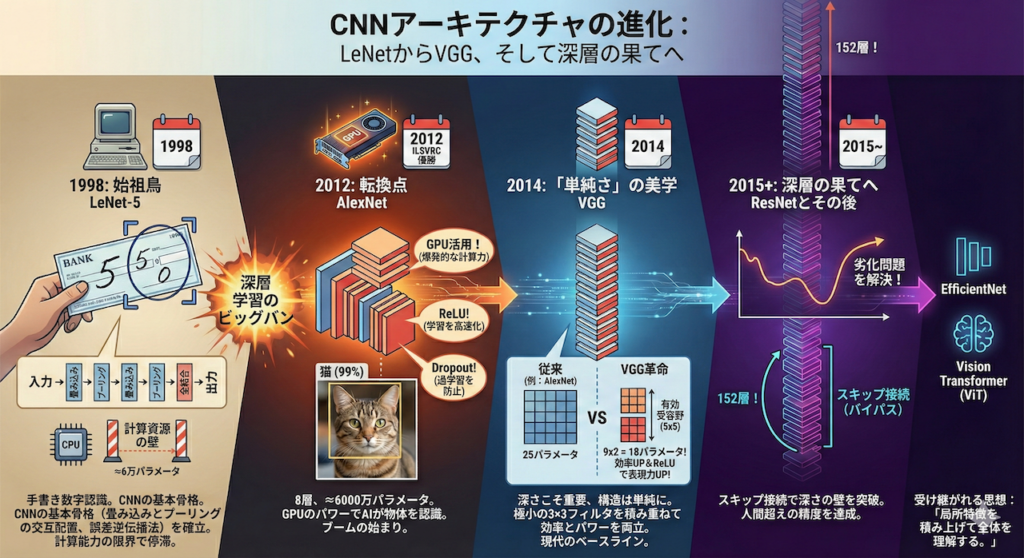
始祖鳥としての「LeNet-5」 (1998)
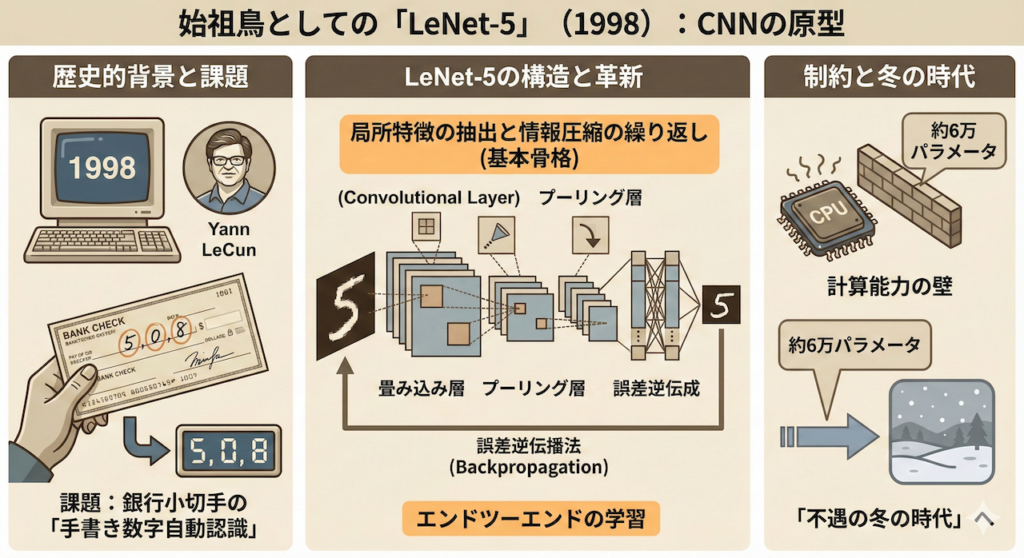
全ての物語はここから始まりました。1998年、現在AI界の巨人として知られるヤン・ルカン(Yann LeCun)博士らが発表した LeNet-5 は、CNNの「原型」を完成させた記念碑的なモデルです (LeCun et al. 1998)。
当時、彼らが挑んでいた課題は「銀行小切手に書かれた手書き数字の自動認識」でした。LeNet-5は、以下の構造を持つことで、このタスクにおいて実用レベルの精度を叩き出しました。
- 畳み込み層とプーリング層の交互配置: 局所特徴の抽出と情報の圧縮を繰り返す、現在のCNNの基本骨格。
- 誤差逆伝播法(Backpropagation)による学習: エンドツーエンドでフィルタの重みを自動調整する仕組み。
しかし、当時のコンピュータの計算能力はあまりに貧弱でした。LeNet-5は約6万個のパラメータしか持っていませんでしたが、これ以上の規模のモデルを学習させることは困難であり、CNNはその後、「不遇の冬の時代」を過ごすことになります。
深層学習のビッグバン:AlexNet (2012)

時計の針を一気に進めましょう。2012年、画像認識コンペティション「ILSVRC」において、ジェフリー・ヒントン教授率いるトロント大学のチームが発表した AlexNet が、2位以下に圧倒的な大差をつけて優勝しました (Krizhevsky et al. 2012)。
これが「ディープラーニング・ブーム」の火付け役となった瞬間です。AlexNetは構造こそLeNetを踏襲していましたが、決定的な違いがありました。
- GPUの活用: グラフィックボードを科学計算に転用し、爆発的な計算力を手に入れた。
- ReLUの採用: 学習を高速化し、勾配消失を防ぐ活性化関数を導入。
- Dropout: ネットワークの一部をランダムに無効化して過学習を防ぐ技術。
これにより、モデルの層数は8層へ、パラメータ数は6000万個へと巨大化しました。AIはついに「猫」を猫として認識する力を手に入れたのです。
「単純さ」の美学:VGG (2014)
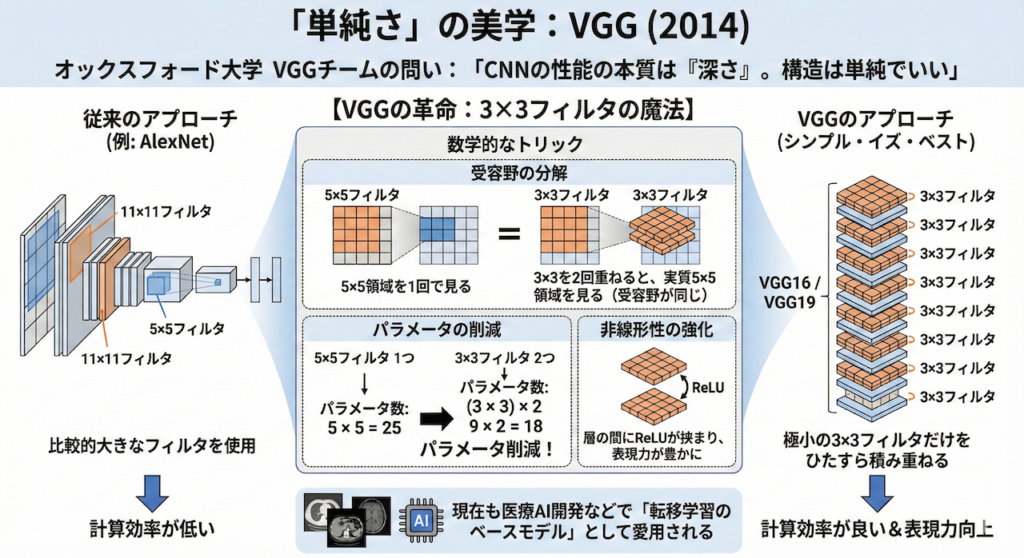
2014年、オックスフォード大学のVGG(Visual Geometry Group)チームは、ある重要な問いを立てました。
「CNNの性能を決める本質は何か? それは『深さ』であり、構造はもっと単純でいいのではないか?」
彼らが開発した VGG16 / VGG19 は、非常にシンプルかつ洗練された設計思想を持っていました (Simonyan & Zisserman 2015)。
【VGGの革命:3×3フィルタの魔法】
それまでのCNN(AlexNetなど)は、\(11 \times 11\) や \(5 \times 5\) といった比較的大きなフィルタを使っていました。しかしVGGは、極小の \(3 \times 3\) フィルタ だけをひたすら積み重ねる戦略を採りました。
なぜ「小さいフィルタ」が良いのでしょうか? ここには数学的なトリックがあります。
- 受容野の分解: \(3 \times 3\) の畳み込みを2回行うと、実質的に \(5 \times 5\) の領域を見ることができます(受容野が同じになる)。
- パラメータの削減:
- \(5 \times 5\) フィルタ1つ: パラメータ数は \(25\)
- \(3 \times 3\) フィルタ2つ: パラメータ数は \(9 \times 2 = 18\)
- 非線形性の強化: 層が増えるたびに活性化関数(ReLU)が挟まるため、表現力が豊かになります。
この「シンプル・イズ・ベスト」な構造は非常に扱いやすく、現在でも転移学習のベースモデルとして、医療AI開発の現場で最も愛されるモデルの一つとなっています。
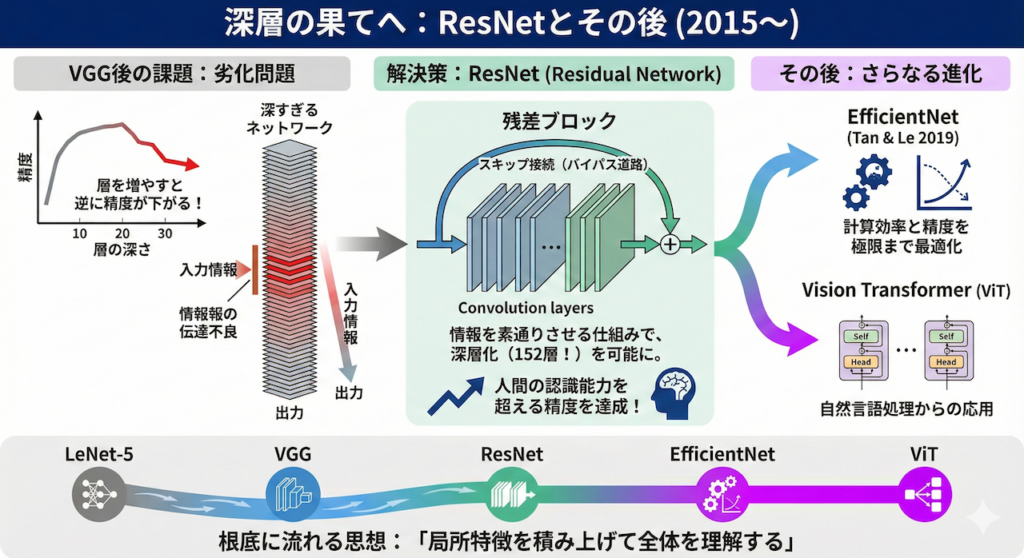
深層の果てへ:ResNetとその後 (2015~)
VGGによって「深ければ深いほど良い」と思われましたが、層を20層、30層と増やすと、逆に精度が下がるという奇妙な現象(劣化問題)が起き始めました。深すぎて、入力側の情報が出力側まで届かなくなってしまったのです。
この壁を突破したのが、2015年の ResNet (Residual Network) です (He et al. 2016)。
ResNetは、「スキップ接続(Skip Connection)」というバイパス道路を設けることで、情報を素通りさせる仕組みを作りました。これにより、152層という驚異的な深さの学習が可能になり、人間の認識能力を超える精度を達成しました。
現在では、計算効率と精度を極限まで最適化した EfficientNet (Tan & Le 2019) や、自然言語処理から輸入された Vision Transformer (ViT) が登場していますが、その根底には、LeNetからVGGへと受け継がれてきた「局所特徴を積み上げて全体を理解する」という思想が脈々と流れています。
3. 医療AI開発の最大の壁:「データ不足」をどう乗り越えるか?
ディープラーニングは「データの量」が性能に直結する技術です。GoogleやMetaが開発する一般的な画像認識AIは、数億枚〜数十億枚という途方もない量の画像で学習しています。
しかし、医療の現場では事情が異なります。特定の希少疾患の画像は、世界中を探しても数百枚しか集まらないことも珍しくありません。また、専門医によるアノテーション(正解ラベル付け)には多大なコストと時間がかかります。
この「少数のデータ(Small Data)」というハンディキャップを克服し、実用的なAIを作るための2つの必須テクニックを紹介します。
A. データ拡張(Data Augmentation):手持ちのデータを「水増し」する技術
データ拡張とは、手元にある画像に対して画像処理を加えることで、擬似的にデータのバリエーションを増やす技術です (Shorten & Khoshgoftaar 2019)。
- 幾何学的変換: 回転、平行移動、拡大縮小、反転(Flip)
- 画質変換: 輝度(明るさ)の変更、コントラスト調整、ノイズ付加
これにより、AIは「画像が少し暗くても」「腫瘍の位置が少しズレていても」、それは同じ病気であると認識できる「頑健性(Robustness)」を獲得します。
【⚠ 医学的な注意点:その変換は「生理学的」に正しいか?】
医療画像でデータ拡張を行う際は、その変換が医学的な意味を変えてしまわないか、細心の注意が必要です。
- 左右反転(Horizontal Flip): 皮膚病変(ダーモスコピー)の画像なら問題ありません。しかし、胸部X線写真を左右反転させると、心臓が右にある「右胸心(Dextrocardia)」という別の病態を作り出してしまいます。AIが「心臓の向き」を学習すべきタスクでは、左右反転は禁忌です。
- 上下反転(Vertical Flip): 病理画像(細胞レベル)では有効ですが、X線やCTのような重力が関係する画像(胸水が下に溜まるなど)では、物理的にあり得ない画像になってしまうため不適切です。
B. 転移学習(Transfer Learning):巨人の肩に乗る
転移学習は、現在の医療AI開発において「必須」と言えるテクニックです。これは、ImageNetのような大規模な一般画像データセット(犬、猫、車など1400万枚以上)で事前に学習させたモデル(Pre-trained Model)を、医療画像用に再利用する手法です (Raghu et al. 2019)。
【なぜ「犬や猫」の学習が「レントゲン」に役立つのか?】
一見、全く関係ないように思えますが、CNNが画像の認識過程で獲得する「視覚能力」には共通点があります。
- 浅い層(共通スキル): 画像のエッジ(輪郭)、曲線、基本的なテクスチャを認識する能力。これは、被写体が猫であろうと腫瘍であろうと共通して必要な能力です。
- 深い層(専門スキル): 「猫の耳」や「肺の結節」といった具体的な対象物を認識する能力。
転移学習では、浅い層の「基礎的な視覚能力」をそのまま借りてきて、深い層だけを医療画像用に微調整(Fine-tuning)します。
【直感的なアナロジー:医学生の教育】
これを人間の教育に例えると、非常に分かりやすくなります。
- ゼロから学習(Scratch): 生まれたばかりの赤ちゃんに、いきなりレントゲンの読み方を教えるようなもの。言葉も形も分からない状態からでは、膨大な時間とデータが必要です。
- 転移学習(Transfer): 「一般教養を修了した大学生」に医学を教えるようなもの。彼らは既に「丸い形」「境界線」といった概念を知っています(基礎能力)。そのため、放射線科の専門教育(Fine-tuning)を短期間受けるだけで、優秀な医師になれるのです。
この手法を使うことで、数百枚〜数千枚程度の少ない医療データでも、驚くほど高性能なAIモデルを構築することが可能になります。
4. 実践:PythonによるCNN構築(胸部X線画像の分類)
それでは、実際にコードを書いてみましょう。今回は教育用データセットとして有名な MedMNIST の PneumoniaMNIST(肺炎の有無を分類するタスク)を使用します。
シナリオ: 胸部X線画像から、肺炎(Pneumonia)か正常(Normal)かを判定するAIを作成する。
以下のコードは、Google Colab等のNotebook環境でそのまま実行可能です。
【実行前の準備】
以下のコードで日本語のグラフを表示させるには、あらかじめターミナルやコマンドプロンプトで pip install japanize-matplotlib medmnist を実行してライブラリをインストールしてください。
# 必要なライブラリのインストール(未インストールの場合)
# !pip install medmnist japanize-matplotlib
import torch # PyTorchのメインモジュールをインポート
import torch.nn as nn # ニューラルネットワーク構築用のモジュールをインポート
import torch.optim as optim # 最適化アルゴリズム用のモジュールをインポート
import torch.utils.data as data # データローダーなどのユーティリティをインポート
import torchvision.transforms as transforms # 画像の前処理用モジュールをインポート
import medmnist # MedMNISTライブラリをインポート
from medmnist import INFO # MedMNISTのデータセット情報をインポート
import matplotlib.pyplot as plt # グラフ描画用ライブラリをインポート
import japanize_matplotlib # matplotlibの日本語化対応ライブラリをインポート
import numpy as np # 数値計算用ライブラリNumPyをインポート
import random # 乱数生成用モジュールをインポート
import os # OS関連機能用モジュールをインポート
# 1. 再現性の確保(乱数シードの固定)
# 実験結果を再現できるように、乱数の種(シード)を固定します。
def set_seed(seed=42):
random.seed(seed) # Python標準の乱数シードを固定
np.random.seed(seed) # NumPyの乱数シードを固定
torch.manual_seed(seed) # PyTorchのCPU用乱数シードを固定
torch.cuda.manual_seed(seed) # PyTorchのGPU用乱数シードを固定
torch.backends.cudnn.deterministic = True # CuDNNの決定論的モードを有効化
set_seed(42) # シード値を42に設定
# デバイスの設定(GPUが使えるならGPUへ、なければCPUを使用)
device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')
print(f"使用デバイス: {device}") # 使用するデバイスを表示
# 2. データセットの準備 (MedMNIST: PneumoniaMNIST)
# データセットの識別子を指定
data_flag = 'pneumoniamnist'
# データセットの情報を取得
info = INFO[data_flag]
n_channels = info['n_channels'] # チャンネル数(グレースケールなので1)
n_classes = len(info['label']) # クラス数(肺炎か否かなので2)
# 前処理の定義(Tensor化と正規化)
# 画像データをPyTorchで扱えるTensor形式に変換し、値を正規化します。
data_transform = transforms.Compose([
transforms.ToTensor(), # 画像をTensor形式に変換
transforms.Normalize(mean=[.5], std=[.5]) # 平均0.5, 標準偏差0.5で正規化
])
# データのダウンロードとロード
# 訓練用データとテスト用データをダウンロードします。
train_dataset = medmnist.PneumoniaMNIST(split='train', transform=data_transform, download=True)
test_dataset = medmnist.PneumoniaMNIST(split='test', transform=data_transform, download=True)
# データローダーの作成(バッチごとにデータを供給する仕組み)
# 学習時にデータを少しずつ(バッチサイズ分)取り出すためのローダーを作成します。
BATCH_SIZE = 64 # バッチサイズを64に設定
train_loader = data.DataLoader(dataset=train_dataset, batch_size=BATCH_SIZE, shuffle=True) # 訓練データローダー(シャッフルあり)
test_loader = data.DataLoader(dataset=test_dataset, batch_size=BATCH_SIZE, shuffle=False) # テストデータローダー(シャッフルなし)
print(f"学習データ数: {len(train_dataset)}, テストデータ数: {len(test_dataset)}") # データ数を表示
# 画像の確認
# データが正しく読み込まれているか、最初の数枚を表示して確認します。
images, labels = next(iter(train_loader)) # 最初のバッチを取得
fig, axes = plt.subplots(1, 5, figsize=(15, 3)) # 5枚の画像を表示する枠を作成
for i in range(5):
img = images[i].squeeze().numpy() # TensorをNumPy配列に変換し、次元を調整
label = "肺炎" if labels[i].item() == 1 else "正常" # ラベルを文字に変換
axes[i].imshow(img, cmap='gray') # 画像をグレースケールで表示
axes[i].set_title(label) # タイトル(正解ラベル)を設定
axes[i].axis('off') # 軸を非表示に
plt.show() # 画像を表示
# 3. シンプルなCNNモデルの定義
# 畳み込み層とプーリング層を組み合わせた基本的なCNNを定義します。
class SimpleCNN(nn.Module):
def __init__(self, in_channels, num_classes):
super(SimpleCNN, self).__init__() # 親クラスの初期化
# 特徴抽出部分 (Convolution -> ReLU -> Pooling)
self.features = nn.Sequential(
# ブロック1: 入力(1ch) -> 出力(16ch)
# カーネルサイズ3x3で畳み込みを行います。
nn.Conv2d(in_channels, 16, kernel_size=3, padding=1), # 畳み込み層
nn.BatchNorm2d(16), # バッチ正規化(学習を安定させる)
nn.ReLU(), # 活性化関数(非線形変換)
nn.MaxPool2d(kernel_size=2, stride=2), # サイズ半減 (28->14)
# ブロック2: 入力(16ch) -> 出力(32ch)
nn.Conv2d(16, 32, kernel_size=3, padding=1), # 畳み込み層
nn.BatchNorm2d(32), # バッチ正規化
nn.ReLU(), # 活性化関数
nn.MaxPool2d(kernel_size=2, stride=2), # サイズ半減 (14->7)
# ブロック3: 入力(32ch) -> 出力(64ch)
nn.Conv2d(32, 64, kernel_size=3, padding=1), # 畳み込み層
nn.BatchNorm2d(64), # バッチ正規化
nn.ReLU(), # 活性化関数
nn.MaxPool2d(kernel_size=2, stride=2) # サイズ半減 (7->3)
)
# 分類部分 (全結合層)
# 抽出された特徴マップを一次元に平坦化し、最終的なクラス分類を行います。
self.classifier = nn.Sequential(
nn.Flatten(), # 特徴マップを平坦化
nn.Linear(64 * 3 * 3, 128), # 特徴マップをベクトル化(入力サイズに合わせて調整)
nn.ReLU(), # 活性化関数
nn.Linear(128, num_classes) # 最終出力層(クラス数へ変換)
)
def forward(self, x):
# 順伝播の定義:特徴抽出 -> 分類
x = self.features(x) # 特徴抽出
x = self.classifier(x) # 分類
return x
# モデルのインスタンス化
# 定義したモデルを作成し、GPU(またはCPU)に転送します。
model = SimpleCNN(in_channels=n_channels, num_classes=n_classes).to(device)
# 4. 学習の実行
# 損失関数と最適化アルゴリズムを定義します。
criterion = nn.CrossEntropyLoss() # 損失関数(分類タスク用、交差エントロピー誤差)
optimizer = optim.Adam(model.parameters(), lr=0.001) # 最適化アルゴリズム(Adam)
epochs = 5 # エポック数(データセットを何周するか)
train_losses = [] # 学習損失を記録するリスト
print("学習開始...")
for epoch in range(epochs):
model.train() # 訓練モードに設定
running_loss = 0.0 # 1エポックごとの損失合計
for inputs, targets in train_loader:
# データをデバイスへ転送
inputs, targets = inputs.to(device), targets.to(device)
targets = targets.squeeze().long() # ラベルの形状調整(1次元配列へ、型をlongへ)
optimizer.zero_grad() # 勾配初期化(前のイテレーションの勾配をリセット)
outputs = model(inputs) # 推論(順伝播)
loss = criterion(outputs, targets) # 誤差計算
loss.backward() # 誤差逆伝播(勾配を計算)
optimizer.step() # パラメータ更新
running_loss += loss.item() # 損失を加算
# 1エポックごとの平均損失を計算・記録
epoch_loss = running_loss / len(train_loader)
train_losses.append(epoch_loss)
print(f"Epoch [{epoch+1}/{epochs}], Loss: {epoch_loss:.4f}")
# 5. 評価
# テストデータを使ってモデルの精度を評価します。
model.eval() # 評価モードに設定
correct = 0 # 正解数
total = 0 # 全データ数
with torch.no_grad(): # 勾配計算なし(メモリ節約・高速化)
for inputs, targets in test_loader:
inputs, targets = inputs.to(device), targets.to(device) # データをデバイスへ転送
targets = targets.squeeze().long() # ラベルの形状調整
outputs = model(inputs) # 推論
_, predicted = torch.max(outputs.data, 1) # 最も確率の高いクラスを予測
total += targets.size(0) # バッチサイズを加算
correct += (predicted == targets).sum().item() # 正解数をカウント
acc = 100 * correct / total # 正解率を計算
print(f"テストデータでの正解率: {acc:.2f}%")
# 学習曲線の表示
# 損失の推移をグラフ化します。
plt.plot(train_losses, label='Training Loss') # 損失のプロット
plt.xlabel('Epoch') # X軸ラベル
plt.ylabel('Loss') # Y軸ラベル
plt.title('学習曲線') # タイトル
plt.legend() # 凡例表示
plt.show() # グラフ表示コードの解説
- MedMNISTの利用: 実際の医療画像(ここでは胸部X線)を、MNIST(手書き数字)のように扱いやすくパッケージ化したライブラリです (Yang et al. 2023)。
nn.Conv2d: これが「フィルタ」の役割を果たします。入力画像の特徴を抽出し、深い層に行くほど抽象的な特徴(線→形→肺の影)を捉えます。nn.MaxPool2d: 情報を圧縮し、計算量を減らしつつ重要な特徴を残します。model.train()/model.eval(): 訓練時と推論時で挙動が変わる層(BatchNormなど)があるため、明示的にモードを切り替えます。
結果
使用デバイス: cuda
100%|██████████| 4.17M/4.17M [00:01<00:00, 3.13MB/s]
学習データ数: 4708, テストデータ数: 624
学習開始...
Epoch [1/5], Loss: 0.1896
Epoch [2/5], Loss: 0.0942
Epoch [3/5], Loss: 0.0845
Epoch [4/5], Loss: 0.0596
Epoch [5/5], Loss: 0.0476
テストデータでの正解率: 87.82%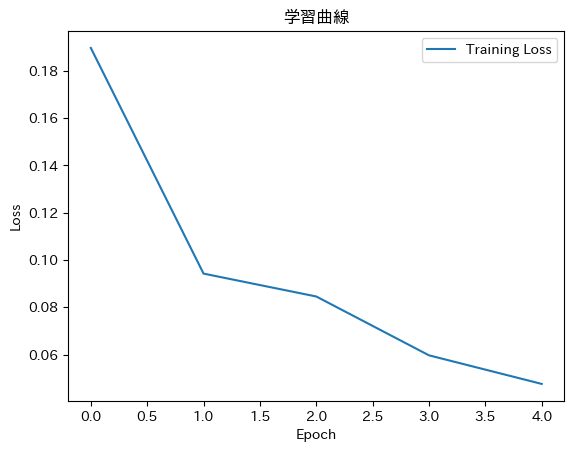
5. 結論:AIの「目」を臨床に活かすために
ここまで、CNNの解剖学から歴史、そしてPythonによる実装までを駆け足で見てきました。最後に、これまでの学びを臨床医としての視点から再統合してみましょう。
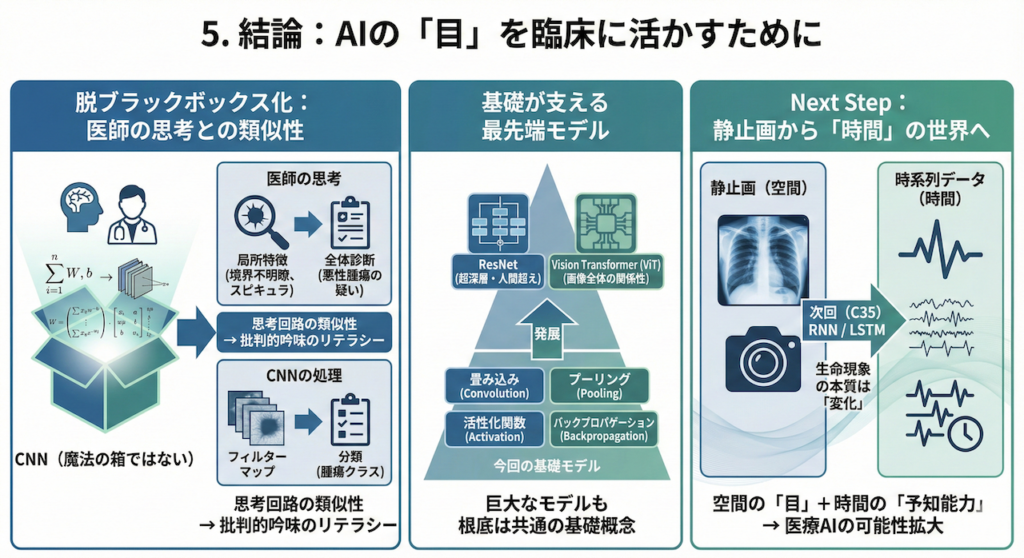
CNNは「魔法の箱」ではない
かつて、ディープラーニングは中身の分からない「ブラックボックス」と揶揄されることがありました。しかし、その内部で行われていることは、決して理解不能な魔法ではありません。
私たちが学んだ通り、CNNは「局所的な特徴(エッジやテクスチャ)」を積み上げ、統合し、より高次の「意味(診断)」へと変換する数理モデルです。これは、医師が診療において行っているプロセスと驚くほど似ています。
- 医師の思考: 「境界不明瞭な陰影がある(局所特徴)」+「スピキュラが見られる(局所特徴)」→「悪性腫瘍の疑い(全体診断)」
- CNNの処理: 「エッジ検出フィルタが反応」+「特定のテクスチャフィルタが反応」→「腫瘍クラスへの分類」
この「思考回路の類似性」を理解していることは、将来AIが提示した診断結果を鵜呑みにせず、批判的に吟味(クリティカル・アプレイザル)する上で極めて重要なリテラシーとなります (Rudin 2019)。
基礎から最先端へ:今回のモデルの位置づけ
今回構築した3層のCNNは、教育用に単純化されたモデルです。実際の臨床研究や製品開発では、より深く複雑なモデルが用いられます。
- ResNet (Residual Network): 100層を超える深さでも学習を可能にし、人間の認識精度を超えたデファクトスタンダード (He et al. 2016)。
- Vision Transformer (ViT): CNNの「局所性」という制約を取り払い、画像全体の関係性を一度に捉える最新鋭のアーキテクチャ (Dosovitskiy et al. 2020)。
しかし、恐れる必要はありません。どんなに巨大なモデルであっても、その根底にあるのは今回学んだ「畳み込み」「プーリング」「活性化関数」「バックプロパゲーション」という基礎概念の組み合わせに過ぎないからです。
Next Step:静止画から「時間」の世界へ
画像診断は医療の重要な柱ですが、生命現象の本質は「変化」にあります。
心臓の鼓動(心電図)、脳の電気活動(脳波)、刻一刻と変動するバイタルサイン。これらは一枚の画像(静止画)ではなく、「時系列データ」として記録されます。
次回(C35)からは、AIに「時間」という次元を教えるための技術、リカレントニューラルネットワーク(RNN)とLSTMの世界へ足を踏み入れます。
空間を認識する「目」に加えて、時間の流れを読む「予知能力」を手に入れたとき、医療AIの可能性はさらに大きく広がります。
参考文献
- He, K. et al. (2016). Deep Residual Learning for Image Recognition. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition (CVPR), 770–778.
- LeCun, Y. et al. (1998). Gradient-based learning applied to document recognition. Proceedings of the IEEE, 86(11), 2278–2324.
- Nair, V. & Hinton, G.E. (2010). Rectified Linear Units Improve Restricted Boltzmann Machines. Proceedings of the 27th International Conference on Machine Learning (ICML), 807–814.
- Raghu, M. et al. (2019). Transfusion: Understanding Transfer Learning for Medical Imaging. Advances in Neural Information Processing Systems (NeurIPS), 32.
- Shorten, C. & Khoshgoftaar, T.M. (2019). A survey on Image Data Augmentation for Deep Learning. Journal of Big Data, 6, Article number: 60.
- Simonyan, K. & Zisserman, A. (2015). Very Deep Convolutional Networks for Large-Scale Image Recognition. International Conference on Learning Representations (ICLR).
- Tan, M. & Le, Q. (2019). EfficientNet: Rethinking Model Scaling for Convolutional Neural Networks. Proceedings of the 36th International Conference on Machine Learning (ICML), 6105–6114.
- Yang, J. et al. (2023). MedMNIST v2: A large-scale lightweight benchmark for 2D and 3D biomedical image classification. Scientific Data, 10, Article number: 41.
- Hubel, D.H. and Wiesel, T.N. (1962). Receptive fields, binocular interaction and functional architecture in the cat’s visual cortex. The Journal of Physiology, 160(1), pp.106–154.
- LeCun, Y. et al. (1998). Gradient-based learning applied to document recognition. Proceedings of the IEEE, 86(11), pp.2278–2324.
- Litjens, G. et al. (2017). A survey on deep learning in medical image analysis. Medical Image Analysis, 42, pp.60–88.
- LeCun, Y. et al. (1998). Gradient-based learning applied to document recognition. Proceedings of the IEEE, 86(11), pp.2278–2324.
- Nair, V. and Hinton, G.E. (2010). Rectified Linear Units Improve Restricted Boltzmann Machines. Proceedings of the 27th International Conference on Machine Learning (ICML), pp.807–814.
- Tan, M. and Le, Q. (2019). EfficientNet: Rethinking Model Scaling for Convolutional Neural Networks. Proceedings of the 36th International Conference on Machine Learning (ICML), pp.6105–6114.
- He, K. et al. (2016). Deep Residual Learning for Image Recognition. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition (CVPR), pp.770–778.
- Krizhevsky, A., Sutskever, I. and Hinton, G.E. (2012). ImageNet Classification with Deep Convolutional Neural Networks. Advances in Neural Information Processing Systems (NeurIPS), 25.
- LeCun, Y. et al. (1998). Gradient-based learning applied to document recognition. Proceedings of the IEEE, 86(11), pp.2278–2324.
- Simonyan, K. and Zisserman, A. (2015). Very Deep Convolutional Networks for Large-Scale Image Recognition. International Conference on Learning Representations (ICLR).
- Dosovitskiy, A. et al. (2020). An Image is Worth 16×16 Words: Transformers for Image Recognition at Scale. International Conference on Learning Representations (ICLR).
- He, K. et al. (2016). Deep Residual Learning for Image Recognition. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition (CVPR), pp.770–778.
- Rudin, C. (2019). Stop explaining black box machine learning models for high stakes decisions and use interpretable models instead. Nature Machine Intelligence, 1(5), pp.206–215.
- Raghu, M. et al. (2019). Transfusion: Understanding Transfer Learning for Medical Imaging. Advances in Neural Information Processing Systems (NeurIPS), 32.
- Shorten, C. and Khoshgoftaar, T.M. (2019). A survey on Image Data Augmentation for Deep Learning. Journal of Big Data, 6, Article number: 60.
※免責事項
本記事は情報提供を目的としたものであり、特定の治療法や医療機器の使用を推奨するものではありません。提供されるサンプルコードは教育目的であり、臨床現場での使用を想定した安全性・有効性の検証は行われていません。健康に関するご懸念やご相談は、必ず専門の医療機関にご相談ください。
ご利用規約(免責事項)
当サイト(以下「本サイト」といいます)をご利用になる前に、本ご利用規約(以下「本規約」といいます)をよくお読みください。本サイトを利用された時点で、利用者は本規約の全ての条項に同意したものとみなします。
第1条(目的と情報の性質)
- 本サイトは、医療分野におけるAI技術に関する一般的な情報提供および技術的な学習機会の提供を唯一の目的とします。
- 本サイトで提供されるすべてのコンテンツ(文章、図表、コード、データセットの紹介等を含みますが、これらに限定されません)は、一般的な学習参考用であり、いかなる場合も医学的な助言、診断、治療、またはこれらに準ずる行為(以下「医行為等」といいます)を提供するものではありません。
- 本サイトのコンテンツは、特定の製品、技術、または治療法の有効性、安全性を保証、推奨、または広告・販売促進するものではありません。紹介する技術には研究開発段階のものが含まれており、その臨床応用には、さらなる研究と国内外の規制当局による正式な承認が別途必要です。
- 本サイトは、情報提供を目的としたものであり、特定の治療法を推奨するものではありません。健康に関するご懸念やご相談は、必ず専門の医療機関にご相談ください。
第2条(法令等の遵守)
利用者は、本サイトの利用にあたり、医師法、医薬品、医療機器等の品質、有効性及び安全性の確保等に関する法律(薬機法)、個人情報の保護に関する法律、医療法、医療広告ガイドライン、その他関連する国内外の全ての法令、条例、規則、および各省庁・学会等が定める最新のガイドライン等を、自らの責任において遵守するものとします。これらの適用判断についても、利用者が自ら関係各所に確認するものとし、本サイトは一切の責任を負いません。
第3条(医療行為における責任)
- 本サイトで紹介するAI技術・手法は、あくまで研究段階の技術的解説であり、実際の臨床現場での診断・治療を代替、補助、または推奨するものでは一切ありません。
- 医行為等に関する最終的な判断、決定、およびそれに伴う一切の責任は、必ず法律上その資格を認められた医療専門家(医師、歯科医師等)が負うものとします。AIによる出力を、資格を有する専門家による独立した検証および判断を経ずに利用することを固く禁じます。
- 本サイトの情報に基づくいかなる行為によって利用者または第三者に損害が生じた場合も、本サイト運営者は一切の責任を負いません。実際の臨床判断に際しては、必ず担当の医療専門家にご相談ください。本サイトの利用によって、利用者と本サイト運営者の間に、医師と患者の関係、またはその他いかなる専門的な関係も成立するものではありません。
第4条(情報の正確性・完全性・有用性)
- 本サイトは、掲載する情報(数値、事例、ソースコード、ライブラリのバージョン等)の正確性、完全性、網羅性、有用性、特定目的への適合性、その他一切の事項について、何ら保証するものではありません。
- 掲載情報は執筆時点のものであり、予告なく変更または削除されることがあります。また、技術の進展、ライブラリの更新等により、情報は古くなる可能性があります。利用者は、必ず自身で公式ドキュメント等の最新情報を確認し、自らの責任で情報を利用するものとします。
第5条(AI生成コンテンツに関する注意事項)
本サイトのコンテンツには、AIによる提案を基に作成された部分が含まれる場合がありますが、公開にあたっては人間による監修・編集を経ています。利用者が生成AI等を用いる際は、ハルシネーション(事実に基づかない情報の生成)やバイアスのリスクが内在することを十分に理解し、その出力を鵜呑みにすることなく、必ず専門家による検証を行うものとします。
第6条(知的財産権)
- 本サイトを構成するすべてのコンテンツに関する著作権、商標権、その他一切の知的財産権は、本サイト運営者または正当な権利を有する第三者に帰属します。
- 本サイトのコンテンツを引用、転載、複製、改変、その他の二次利用を行う場合は、著作権法その他関連法規を遵守し、必ず出典を明記するとともに、権利者の許諾を得るなど、適切な手続きを自らの責任で行うものとします。
第7条(プライバシー・倫理)
本サイトで紹介または言及されるデータセット等を利用する場合、利用者は当該データセットに付随するライセンス条件および研究倫理指針を厳格に遵守し、個人情報の匿名化や同意取得の確認など、適用される法規制に基づき必要とされるすべての措置を、自らの責任において講じるものとします。
第8条(利用環境)
本サイトで紹介するソースコードやライブラリは、執筆時点で特定のバージョンおよび実行環境(OS、ハードウェア、依存パッケージ等)を前提としています。利用者の環境における動作を保証するものではなく、互換性の問題等に起因するいかなる不利益・損害についても、本サイト運営者は責任を負いません。
第9条(免責事項)
- 本サイト運営者は、利用者が本サイトを利用したこと、または利用できなかったことによって生じる一切の損害(直接損害、間接損害、付随的損害、特別損害、懲罰的損害、逸失利益、データの消失、プログラムの毀損等を含みますが、これらに限定されません)について、その原因の如何を問わず、一切の法的責任を負わないものとします。
- 本サイトの利用は、学習および研究目的に限定されるものとし、それ以外の目的での利用はご遠慮ください。
- 本サイトの利用に関連して、利用者と第三者との間で紛争が生じた場合、利用者は自らの費用と責任においてこれを解決するものとし、本サイト運営者に一切の迷惑または損害を与えないものとします。
- 本サイト運営者は、いつでも予告なく本サイトの運営を中断、中止、または内容を変更できるものとし、これによって利用者に生じたいかなる損害についても責任を負いません。
第10条(規約の変更)
本サイト運営者は、必要と判断した場合、利用者の承諾を得ることなく、いつでも本規約を変更することができます。変更後の規約は、本サイト上に掲載された時点で効力を生じるものとし、利用者は変更後の規約に拘束されるものとします。
第11条(準拠法および合意管轄)
本規約の解釈にあたっては、日本法を準拠法とします。本サイトの利用および本規約に関連して生じる一切の紛争については、東京地方裁判所を第一審の専属的合意管轄裁判所とします。
For J³, may joy follow you.