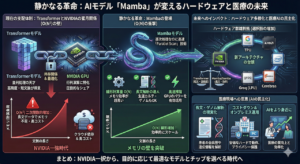
Gitはコードのための「電子カルテ」です。変更履歴の追跡(Git)、チームによる査読(GitHub)、そして再現性の担保(環境固定)を組み合わせることで、医療安全レベルのAI開発を実現します。
ファイル名を変えずに、データベースで履歴を管理します。「誰が・いつ・なぜ」変更したかを記録し、いつでも過去の状態へ戻せる安全性を確保します。
本番コード(標準治療)に影響を与えず、ブランチ(治験環境)で新機能を実験。検証完了後にプルリクエスト(査読)を経て安全に統合します。
Gitでのコード管理に加え、データの不変性(Read Only)とドキュメント(手順書)の整備が不可欠です。これら3つが揃って初めて科学的再現性が成立します。
はじめに:科学における「再現性の危機」とコードの監査記録
現代の医科学研究において、「再現性の危機(Reproducibility Crisis)」は看過できない課題となっています。Nature誌が実施した1,500人以上の研究者を対象とした調査では、過半数が「再現性の危機は深刻である」と考え、70%以上の研究者が他の研究者の実験結果を再現しようとして失敗した経験があると回答しています (Baker 2016)。
医療AI開発において、この問題はさらに複雑化します。解析コードのわずかなパラメータ変更、前処理手順の修正、ライブラリのバージョン差異によって、モデルの予測精度や統計結果は劇的に変化します。もし、あなたが数ヶ月前の解析結果を「完全に」再現できないとしたら、そのAIモデルは臨床応用における信頼性(Reliability)と説明責任(Accountability)を欠いていると言わざるを得ません (Ioannidis 2005)。
本稿では、ソフトウェア工学の標準ツールである Git(ギット) と GitHub(ギットハブ) を、医療における「電子カルテシステム」や「監査証跡(Audit Trail)」のアナロジーを用いて解説します。これらは単なるプログラム管理ツールではなく、医療AI研究の透明性を担保し、過去のあらゆる時点の状態へ遡及可能にするための必須技術です (The Turing Way Community 2022)。
1. Gitとは何か:コードのための電子カルテ
1.1 バージョン管理システム(VCS)の役割
臨床現場において、紙カルテから電子カルテ(EHR)への移行がもたらした最大の恩恵の一つは「履歴の追跡可能性」です。誰が、いつ、どの記載を修正したか、修正前の値は何だったかが記録されます。
Git は、まさに「ソースコードのための電子カルテ」です。分散型バージョン管理システム(Distributed Version Control System)と呼ばれ、ファイルの変更履歴をすべて記録します。
- 従来のファイル管理(ファイル名管理):
analysis_v1.py,analysis_final.py,analysis_final_rev2.pyのような管理は、臨床で言えば「付箋紙で指示を書き換える」ようなものであり、ミスの温床となります。 - Gitによる管理: ファイル名は常に
analysis.pyのままです。Gitが背後で「どの時点のファイルか」という時系列データを厳密に管理します。
1.2 医療プロセスとの対応関係
Gitの基本概念は、以下のように医療プロセスに対応付けると直感的に理解できます。
| Gitの用語 | 医療プロセスのアナロジー | 機能・役割 |
|---|---|---|
| リポジトリ (Repository) | 患者カルテ(一冊分) | プロジェクト全体のファイルと変更履歴の保管場所。 |
| コミット (Commit) | カルテの確定・署名 | 変更内容を記録し、確定させる行為。タイムスタンプとIDが付与される。 |
| ステージング (Staging) | 回診時のメモ・下書き | 確定(コミット)する前に、変更内容を確認・選別する段階。 |
| ブランチ (Branch) | 鑑別診断の仮説検証 | 本流の治療方針に影響を与えず、別の可能性(新機能)を試す並行世界。 |
| マージ (Merge) | 治療方針の統合 | 検証された仮説(ブランチ)を、本流の治療計画(メイン)に統合する。 |
2. Gitの導入と「清潔野」の確保(セットアップ)
医療AI開発を始めるにあたり、まずは環境を整備します。ここでは、自身のコンピュータ(ローカル環境)にGitを導入し、ユーザー情報を登録します。これは電子カルテにおける「ユーザーアカウント作成」に相当し、誰が行った変更かを明確にするために不可欠です。
2.1 インストールと初期設定
※ 多くのMacやLinux環境には標準でGitがインストールされています。Windowsの場合は「Git for Windows」を利用するのが一般的です。
ターミナル(またはコマンドプロンプト)で以下のコマンドを実行し、自身の署名情報を登録します。
# ユーザー名の登録(論文の著者名に相当)
git config --global user.name "Taro Yamada"
# メールアドレスの登録(連絡先に相当)
git config --global user.email "taro.yamada@university-hosp.ac.jp"
2.2 リポジトリの作成(患者登録)
プロジェクトフォルダを作成し、そこをGitの管理下(リポジトリ)として初期化します。
# プロジェクトフォルダの作成と移動
mkdir clinical_ai_project
cd clinical_ai_project
# Gitリポジトリとして初期化(カルテの新規作成)
git init
このコマンドにより、隠しフォルダ .git が生成され、ここに変更履歴データベースが構築され始めます。
3. 基本的なワークフロー:経過記録の作成
コードを書き、変更を記録するプロセスは、「執筆(Coding)」→「確認(Staging)」→「確定(Commit)」の3段階で進みます。
3.1 変更の記録(コミット)
例えば、main.py という解析スクリプトを作成したとします。これを記録する手順は以下の通りです。
# 1. ステージングエリアに追加(カルテの下書き保存・確認)
git add main.py
# 2. コミット(カルテの確定・署名)
# -m オプションで、何をしたかのメッセージ(経過記録)を残す
git commit -m "Add initial analysis script for HbA1c prediction"
ここで重要なのは、コミットメッセージです。「修正」や「更新」といった曖昧な言葉ではなく、「HbA1c予測のための初期スクリプトを追加」のように、「なぜその変更を行ったか」という臨床的・技術的意図を明記することが、将来の再現性のために求められます (Wilson et al. 2017)。
3.2 履歴の確認(ログ参照)
過去の「診療記録」を確認するには git log を使用します。
git logこれにより、誰が、いつ、どのような意図で変更を加えたかが一覧表示されます。必要であれば、過去の任意のコミット時点の状態へ、コード全体を「巻き戻す(Checkout/Revert)」ことが可能です。
3.3 個人情報保護と .gitignore
医療AI開発において極めて重要なのが、「患者データ(個人情報)をGitに記録しない」 という鉄則です。Gitは履歴を永続的に保持するため、一度でも個人情報を含むCSVなどをコミットしてしまうと、履歴から完全に消去するのは困難です。
そこで、.gitignore という特殊な設定ファイルを作成し、管理対象外とするファイルを指定します。
.gitignore ファイルの記述例:
# 患者データや機微情報(絶対にコミットしない)
data/raw/*.csv
data/processed/*.xlsx
# 仮想環境(環境ごとに再構築するため含めない)
.venv/
env/
# Pythonのキャッシュファイル
__pycache__/
*.pyc
これにより、誤って機微なデータをリポジトリに公開してしまうリスクを物理的に遮断します。ただし、GDPRや日本の個人情報保護法(APPI)への完全な準拠は、技術的措置だけでなく、組織的なデータ管理規定やアクセス制御と組み合わせて初めて達成される点に留意が必要です (Personal Information Protection Commission 2022; European Parliament 2016)。
4. ブランチとマージ:仮説検証の安全な実施
臨床研究において、標準治療(Standard of Care)を継続しつつ、一部の対象群で新規治療法の探索を行うように、Gitでは ブランチ(Branch) を使用します。
mainブランチ: 常に動作が保証された、本番用のコード(標準治療)。feature/xxxブランチ: 新しいアルゴリズムや前処理を試すための作業用ブランチ(治験/探索的治療)。
ワークフロー
- 分岐:
git switch -c feature/new-modelで新しいブランチを作成し、移動する。 - 実装: 新しいAIモデルを実装し、コミットを重ねる。この間、
mainブランチ(本番コード)は影響を受けない。 - 検証: 新モデルの精度が良いことが確認されたら、
mainブランチに戻る。 - 統合(マージ):
git merge feature/new-modelで、成果を本番コードに取り込む。
この仕組みにより、実験的なコードでシステム全体を破損させるリスクを回避しながら、安全に開発を進めることができます。
5. GitHubとプルリクエスト:チーム医療と査読
GitHub は、Gitリポジトリをクラウド上でホスティングするサービスです。これは、単なるバックアップ(保管庫)ではなく、「チームによるコードレビュー(査読)」 のプラットフォームとして機能します。
5.1 リモートリポジトリへのプッシュ
ローカルで行った変更履歴を、GitHub上のリモートリポジトリへ送信(アップロード)します。
# リモートリポジトリの登録(初回のみ)
git remote add origin https://github.com/username/repo-name.git
# 変更履歴の送信
git push -u origin main
5.2 プルリクエスト(Pull Request: PR)
チーム開発において、変更をいきなり本番環境(main)に統合することはリスクがあります。そこで プルリクエスト(Pull Request) という手続きを踏みます。
- 開発者は、自分のブランチでの作業完了後、「この変更を
mainに取り込んでください」とリクエスト(PR)を送る。 - レビュアー(他の医師やエンジニア) は、GitHub上でコードの差分を確認する。
- 問題があればコメントで修正を指示し、承認(Approve)された場合のみ統合(Merge)される。
このプロセスは、医学論文における 「査読(Peer Review)」 や、臨床現場における 「カンファレンス」 と同義です。コードの品質、ロジックの正当性、セキュリティリスクを複数の目でチェックすることで、医療AIとしての質を担保します。
6. 実践:再現性を意識したPythonコード
Gitで管理することを前提とした、解析コードの例を示します。ここでは、乱数シードを固定し、ライブラリのバージョンに依存せず結果が再現されるような記述を行います。
【実行前の準備】
以下のコードでグラフ内の日本語を正しく表示させるには、あらかじめターミナルやコマンドプロンプトで japanize-matplotlib をインストールしてください。
pip install japanize-matplotlibPythonサンプルコード:糖尿病データセットの可視化とバージョン管理
ここでは、scikit-learnに付属する糖尿病データセットを用います。これは実在する442人の糖尿病患者のベースライン情報をもとに作成されたものですが、研究用に匿名化・標準化されたデータセットであり、個人特定のリスクはありません (scikit-learn developers 2024)。
🚀 実際に試せるコード (Copy & Run)
以下のコードは実際にローカル環境や Google Colab で動作します。
pip install pandas matplotlib scikit-learn japanize-matplotlib
import pandas as pd
import matplotlib.pyplot as plt
import japanize_matplotlib
import numpy as np
from sklearn.datasets import load_diabetes
RANDOM_SEED = 42
np.random.seed(RANDOM_SEED)
def load_and_preprocess_data():
diabetes = load_diabetes()
df = pd.DataFrame(diabetes.data, columns=diabetes.feature_names)
df['target'] = diabetes.target
return df
def plot_bmi_vs_progression(df, save_path="bmi_progression.png"):
plt.figure(figsize=(10, 6))
plt.scatter(df['bmi'], df['target'], alpha=0.6, c='#1f77b4', label='Data')
plt.title('BMIと糖尿病進行度の関係 (n=442)', fontsize=15)
plt.xlabel('BMI (Standardized)', fontsize=12)
plt.ylabel('Disease Progression', fontsize=12)
plt.grid(True, linestyle='--', alpha=0.7)
plt.legend()
plt.savefig(save_path)
print(f"Saved: {save_path}")
plt.close()
if __name__ == "__main__":
df_patient = load_and_preprocess_data()
print(df_patient[['bmi', 'target']].describe())
plot_bmi_vs_progression(df_patient)
import pandas as pd
import matplotlib.pyplot as plt
import japanize_matplotlib # 日本語表示用ライブラリ
import numpy as np
from sklearn.datasets import load_diabetes
# ---------------------------------------------------------
# 再現性のためのシード固定
# ---------------------------------------------------------
# 乱数シードを固定することで、いつ誰が実行しても同じデータ分割・結果になるようにする
RANDOM_SEED = 42
np.random.seed(RANDOM_SEED)
def load_and_preprocess_data():
"""
データの読み込みと前処理を行う関数
実務ではここでSQLクエリやCSV読み込みを行う
"""
# scikit-learn付属の糖尿病患者データセット(匿名化済み研究用データ)を使用
diabetes = load_diabetes()
df = pd.DataFrame(diabetes.data, columns=diabetes.feature_names)
df['target'] = diabetes.target # 目的変数(1年後の疾患進行度)
# BMIと進行度の関係を見るためにデータを整形
return df
def plot_bmi_vs_progression(df, save_path="bmi_progression.png"):
"""
BMIと疾患進行度の散布図を描画して保存する関数
"""
plt.figure(figsize=(10, 6))
# 散布図の描画
plt.scatter(df['bmi'], df['target'], alpha=0.6, c='#1f77b4', label='患者データ')
# グラフの装飾(日本語対応)
plt.title('BMIと糖尿病進行度の関係 (n=442)', fontsize=15)
plt.xlabel('BMI (標準化値)', fontsize=12)
plt.ylabel('1年後の疾患進行度指標', fontsize=12)
plt.grid(True, linestyle='--', alpha=0.7)
plt.legend()
# 画像として保存(Gitには画像だけでなく、この生成コード自体を管理する)
plt.savefig(save_path)
print(f"グラフを保存しました: {save_path}")
plt.close()
if __name__ == "__main__":
# 1. データ読み込み
df_patient = load_and_preprocess_data()
# 2. 基礎統計量の表示(ログとして残す価値がある)
print("--- データセット統計量 ---")
print(df_patient[['bmi', 'target']].describe())
# 3. 可視化の実行
plot_bmi_vs_progression(df_patient)
コード管理のポイント
上記のコードをGitで管理する場合、重要なのは出力された bmi_progression.png だけでなく、「その画像を生成したコード(main.py)」の履歴 です。
例えば、「散布図の色を青から赤に変えた」「タイトルの文言を修正した」といった変更をコミットとして記録することで、論文投稿時に「図3を出力した正確なコードのバージョン」を特定することが可能になります。
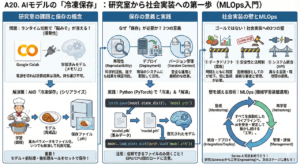
7. 再現性を支える3つのレイヤー:コードだけでは足りない理由
「Gitを使っているから再現性は完璧だ」と考えるのは、外科医が「メスを消毒したから手術は成功する」と考えるのに似ています。道具(Git)は重要ですが、それだけでは十分ではありません。
医療AI研究において、第三者が検証可能なレベルの「完全な再現性」を担保するには、以下の3つのレイヤーがすべて整合性を保って管理されている必要があります (Wilkinson et al. 2016; McDermott et al. 2021)。
7.1 医療AI再現性の階層構造
Gitが直接管理できるのは、主に「レイヤー2」です。しかし、レイヤー1(データ)が破損していたり、レイヤー3(手順)が不明瞭であれば、どんなに優れたコードも無意味になります。
[レイヤー3:コンテキスト(人間への説明責任)]
│ 目的:暗黙知の排除とプロトコルの明文化
├─ 論文 / プレプリント (Scientific Context)
├─ README.md (プロジェクトの「取り扱い説明書」)
└─ 分析手順書 (Standard Operating Procedures: SOPs)
[レイヤー2:ロジックと環境(Git/Dockerの守備範囲)]
│ 目的:計算処理の完全な決定性担保
├─ Git / GitHub (ソースコードの時系列管理)
├─ requirements.txt / environment.yml (ライブラリ依存関係の固定)
└─ Docker / コンテナ (OSレベルの環境凍結)
[レイヤー1:データ(不動の基盤)]
│ 目的:入力の不変性とアクセス制御の両立
├─ Raw Data (生データ:読み取り専用・ハッシュ管理・厳格なアクセス制御)
├─ Processed Data (中間データ:再生成可能な形式)
└─ Synthetic Data / De-identified Data (公開・共有可能な派生データ)
7.2 各レイヤーの役割とリスク
レイヤー1:データ(Data)
最も基盤となる層です。医療データは個人情報保護の観点から厳格なアクセス制御(GDPR, APPI等)が求められますが、同時に科学的には「不変性(Immutability)」が求められます。「1年前の解析に使ったCSVファイル」が上書き保存されて値が変わっていれば、再現は不可能です。生データは「Read Only(読み取り専用)」として保存し、ハッシュ値(データの指紋)で同一性を管理することが推奨されます。
レイヤー2:コード・環境(Code & Environment)
ここでGitが活躍します。しかし、コードだけでなく「実行環境」もセットで管理する必要があります。「私のPCでは動いた(It works on my machine)」という言い訳は、科学の世界では通用しません。requirements.txt や Docker を用いて、ライブラリのバージョンやOS環境ごと「冷凍保存」するように定義します。
レイヤー3:ドキュメント(Documentation)
最上位の層です。どの順序でスクリプトを実行するのか、パラメータの意味は何か、なぜそのアルゴリズムを選んだのか。これら「研究者の頭の中にある暗黙知」を文章化します。優れた README ファイルは、未来の自分自身、そしてあなたの研究を検証しようとする他の研究者への手紙です。
これら3つのレイヤーが揃って初めて、FAIR原則(Findable, Accessible, Interoperable, Reusable)を満たす、質の高い医療AI研究と言えます (Wilkinson et al. 2016)。
おわりに:未来の医療への責任
医療AIにおけるプログラミングは、単なる計算処理の記述ではありません。それは、将来の患者に対する診断や治療方針に影響を与えうる「医療行為の一部」です。
Git と GitHub を用いて履歴を管理し、再現性を担保することは、研究の透明性を高めるだけでなく、開発者であるあなた自身と、そのAIによって恩恵を受ける患者を守るための倫理的責務(Ethical Obligation)でもあります (Topol 2019)。
適切なツールを用いて「時を戻せる」環境を構築し、科学的に堅牢な医療AI開発の第一歩を踏み出しましょう。
参考文献
- Baker, M. (2016). 1,500 scientists lift the lid on reproducibility. Nature, 533(7604), pp. 452–454.
- Beam, A.L. and Kohane, I.S. (2018). Big data and machine learning in health care. JAMA, 319(13), pp. 1317–1318.
- Chacon, S. and Straub, B. (2014). Pro Git. 2nd edn. Apress.
- European Parliament and Council. (2016). Regulation (EU) 2016/679 (General Data Protection Regulation). Official Journal of the European Union, L119, pp. 1–88.
- Ioannidis, J.P.A. (2005). Why most published research findings are false. PLoS Medicine, 2(8), e124.
- 個人情報保護委員会 (2022). 医療・介護関係事業者における個人情報の適切な取扱いのためのガイダンス. 個人情報保護委員会.
- McDermott, M.B.A. et al. (2021). Reproducibility in machine learning for health. Nature Communications, 12, p. 2454.
- scikit-learn developers (2024). Diabetes dataset. scikit-learn documentation, Version 1.5.1.
- The Turing Way Community (2022). The Turing Way: A handbook for reproducible, ethical and collaborative data science. Zenodo.
- Topol, E.J. (2019). High-performance medicine: the convergence of human and artificial intelligence. Nature Medicine, 25(1), pp. 44–56.
- Wilkinson, M.D. et al. (2016). The FAIR Guiding Principles for scientific data management and stewardship. Scientific Data, 3, p. 160018.
- Wilson, G. et al. (2017). Good enough practices in scientific computing. PLoS Computational Biology, 13(6), e1005510.
※本記事は情報提供を目的としたものであり、特定の治療法を推奨するものではありません。健康に関するご懸念やご相談は、必ず専門の医療機関にご相談ください。
※紹介しているコードやツールは教育目的の例示であり、臨床環境での動作を保証するものではありません。実際の医療データを取り扱う際は、所属機関の倫理規定および関連法規(個人情報保護法、次世代医療基盤法など)を遵守してください。
※本記事は法律的助言を提供するものではなく、具体的な法的判断が必要な場合は、必ず専門家にご相談ください。
ご利用規約(免責事項)
当サイト(以下「本サイト」といいます)をご利用になる前に、本ご利用規約(以下「本規約」といいます)をよくお読みください。本サイトを利用された時点で、利用者は本規約の全ての条項に同意したものとみなします。
第1条(目的と情報の性質)
- 本サイトは、医療分野におけるAI技術に関する一般的な情報提供および技術的な学習機会の提供を唯一の目的とします。
- 本サイトで提供されるすべてのコンテンツ(文章、図表、コード、データセットの紹介等を含みますが、これらに限定されません)は、一般的な学習参考用であり、いかなる場合も医学的な助言、診断、治療、またはこれらに準ずる行為(以下「医行為等」といいます)を提供するものではありません。
- 本サイトのコンテンツは、特定の製品、技術、または治療法の有効性、安全性を保証、推奨、または広告・販売促進するものではありません。紹介する技術には研究開発段階のものが含まれており、その臨床応用には、さらなる研究と国内外の規制当局による正式な承認が別途必要です。
- 本サイトは、情報提供を目的としたものであり、特定の治療法を推奨するものではありません。健康に関するご懸念やご相談は、必ず専門の医療機関にご相談ください。
第2条(法令等の遵守)
利用者は、本サイトの利用にあたり、医師法、医薬品、医療機器等の品質、有効性及び安全性の確保等に関する法律(薬機法)、個人情報の保護に関する法律、医療法、医療広告ガイドライン、その他関連する国内外の全ての法令、条例、規則、および各省庁・学会等が定める最新のガイドライン等を、自らの責任において遵守するものとします。これらの適用判断についても、利用者が自ら関係各所に確認するものとし、本サイトは一切の責任を負いません。
第3条(医療行為における責任)
- 本サイトで紹介するAI技術・手法は、あくまで研究段階の技術的解説であり、実際の臨床現場での診断・治療を代替、補助、または推奨するものでは一切ありません。
- 医行為等に関する最終的な判断、決定、およびそれに伴う一切の責任は、必ず法律上その資格を認められた医療専門家(医師、歯科医師等)が負うものとします。AIによる出力を、資格を有する専門家による独立した検証および判断を経ずに利用することを固く禁じます。
- 本サイトの情報に基づくいかなる行為によって利用者または第三者に損害が生じた場合も、本サイト運営者は一切の責任を負いません。実際の臨床判断に際しては、必ず担当の医療専門家にご相談ください。本サイトの利用によって、利用者と本サイト運営者の間に、医師と患者の関係、またはその他いかなる専門的な関係も成立するものではありません。
第4条(情報の正確性・完全性・有用性)
- 本サイトは、掲載する情報(数値、事例、ソースコード、ライブラリのバージョン等)の正確性、完全性、網羅性、有用性、特定目的への適合性、その他一切の事項について、何ら保証するものではありません。
- 掲載情報は執筆時点のものであり、予告なく変更または削除されることがあります。また、技術の進展、ライブラリの更新等により、情報は古くなる可能性があります。利用者は、必ず自身で公式ドキュメント等の最新情報を確認し、自らの責任で情報を利用するものとします。
第5条(AI生成コンテンツに関する注意事項)
本サイトのコンテンツには、AIによる提案を基に作成された部分が含まれる場合がありますが、公開にあたっては人間による監修・編集を経ています。利用者が生成AI等を用いる際は、ハルシネーション(事実に基づかない情報の生成)やバイアスのリスクが内在することを十分に理解し、その出力を鵜呑みにすることなく、必ず専門家による検証を行うものとします。
第6条(知的財産権)
- 本サイトを構成するすべてのコンテンツに関する著作権、商標権、その他一切の知的財産権は、本サイト運営者または正当な権利を有する第三者に帰属します。
- 本サイトのコンテンツを引用、転載、複製、改変、その他の二次利用を行う場合は、著作権法その他関連法規を遵守し、必ず出典を明記するとともに、権利者の許諾を得るなど、適切な手続きを自らの責任で行うものとします。
第7条(プライバシー・倫理)
本サイトで紹介または言及されるデータセット等を利用する場合、利用者は当該データセットに付随するライセンス条件および研究倫理指針を厳格に遵守し、個人情報の匿名化や同意取得の確認など、適用される法規制に基づき必要とされるすべての措置を、自らの責任において講じるものとします。
第8条(利用環境)
本サイトで紹介するソースコードやライブラリは、執筆時点で特定のバージョンおよび実行環境(OS、ハードウェア、依存パッケージ等)を前提としています。利用者の環境における動作を保証するものではなく、互換性の問題等に起因するいかなる不利益・損害についても、本サイト運営者は責任を負いません。
第9条(免責事項)
- 本サイト運営者は、利用者が本サイトを利用したこと、または利用できなかったことによって生じる一切の損害(直接損害、間接損害、付随的損害、特別損害、懲罰的損害、逸失利益、データの消失、プログラムの毀損等を含みますが、これらに限定されません)について、その原因の如何を問わず、一切の法的責任を負わないものとします。
- 本サイトの利用は、学習および研究目的に限定されるものとし、それ以外の目的での利用はご遠慮ください。
- 本サイトの利用に関連して、利用者と第三者との間で紛争が生じた場合、利用者は自らの費用と責任においてこれを解決するものとし、本サイト運営者に一切の迷惑または損害を与えないものとします。
- 本サイト運営者は、いつでも予告なく本サイトの運営を中断、中止、または内容を変更できるものとし、これによって利用者に生じたいかなる損害についても責任を負いません。
第10条(規約の変更)
本サイト運営者は、必要と判断した場合、利用者の承諾を得ることなく、いつでも本規約を変更することができます。変更後の規約は、本サイト上に掲載された時点で効力を生じるものとし、利用者は変更後の規約に拘束されるものとします。
第11条(準拠法および合意管轄)
本規約の解釈にあたっては、日本法を準拠法とします。本サイトの利用および本規約に関連して生じる一切の紛争については、東京地方裁判所を第一審の専属的合意管轄裁判所とします。
For J³, may joy follow you.